Phân tích họ gene mã hóa β-amylase ở cây sắn (Manihot esculenta Crantz) bằng phương pháp tin sinh học
Bạn đang xem bản rút gọn của tài liệu. Xem và tải ngay bản đầy đủ của tài liệu tại đây (527.12 KB, 9 trang )
KHOA HỌC TỰ NHIÊN
Phân tích họ gene mã hóa β-amylase
Ở CÂY SẮN (Manihot esculenta Crantz)
BẰNG PHƯƠNG PHÁP TIN SINH HỌC
Cao Phi Bằng
Khoa Khoa học Tự nhiên – Trường Đại học Hùng Vương
Nhận bài ngày 10/11/2017, Phản biện xong ngày 25/11/2017, Duyệt đăng ngày 26/11/2017
TÓM TẮT
C
ác beta-amylase (β-amylase, EC 3.2.1.2) thuộc họ glycosyl hydrolase 14 có chức
năng phân cắt các liên kết (1,4)-α-D-glycosidic trong các phân tử tinh bột, giải
phóng các maltose từ đầu không khử của chuỗi. Sử dụng phương pháp tin sinh học,
tổng số 10 gene mã hóa β-amylase đã được xác định trong hệ gene của cây sắn. Các
gene β-amylase của cây sắn phân bố trên 5 trong tổng số 18 nhiễm sắc thể. Dựa trên
kết quả phân tích cây phả hệ, các β-amylase của cây sắn được xếp vào bốn phân họ
khác nhau, gồm phân họ I, II, III và IV. Các gene này mã hóa không liên tục với số lượng
intron thay đổi theo phân họ và theo từng gene trong phân họ. Các protein suy diễn
của chúng có mức tương đồng khá cao khi so với các β-amylase cùng phân họ của cây
Arabidopsis. Các protein β-amylase của cây sắn có điểm đẳng điện (pI) dao động từ
5,44 tới 8,92. Tất cả các β-amylase của cây sắn đều mang vùng bảo thủ của họ enzyme
glycosyl hydrolase 14 và hầu hết trong số chúng có mang các amino acid giữ vai trò
quan trọng đối với chức năng enzyme. Ngoài ra, MeBAM5 và MeBAM10 còn chứa
đoạn amino acid giống với vùng bảo thủ của tác nhân điều hòa phiên mã chống chịu
với Brassinazole ở đầu amin.
Từ khóa: β-amylase, cây sắn, cây phả hệ, đặc trưng của gene, tin sinh học.
1.Đặt vấn đề
Cây sắn (Manihot esculenta Crantz) là cây
lương thực quan trọng thứ 6 trên thế giới
chỉ sau cây lúa mì, cây lúa gạo, cây ngô, cây
khoai tây và cây lúa mạch. Cây sắn có nguồn
gốc từ Nam Mỹ thuộc họ Euphorbiaceae
này hiện được trồng khắp vùng nhiệt đới và
cận nhiệt đới châu Phi, châu Á và châu Mỹ,
trong đó có Việt Nam [10]. Củ sắn là nguồn
cung cấp lương thực cho khoảng 800 triệu
người trên toàn cầu, mặc dù tất cả các bộ
phận của cây sắn đều có thể sử dụng [3]. Củ
sắn có hàm lượng tinh bột cao (20-40%), là
một nguồn năng lượng tốt cho nhu cầu của
Tạp chí Khoa học & Công nghệ số 4 (9) – 2017 49
KHOA HỌC TỰ NHIÊN
con người và đặc biệt là trong công nghiệp
nhiên liệu sinh học. Vì là cây có vai trò lớn
đối với con người nên hệ gene của cây sắn
đã được giải trình tự [1; 17], là cơ sở cho các
nghiên cứu về cấu trúc, chức năng và tiến
hóa của gene, công cụ hữu hiệu trong chọn
tạo giống loài cây này.
Các beta-amylase (β-amylase, EC 3.2.1.2)
thuộc họ glycosyl hydrolase 14, xúc tác phản
ứng thủy phân các phân tử amylose và liên
kết (1,4)-α-D-glycosidic trong các phân tử
tinh bột, glycogene và maltooligosaccharide
[16]. β-amylase có ở thực vật bậc cao và
một số vi sinh vật. β-amylase được tích lũy
nhiều trong hạt nảy mầm và trong một số
mô chịu tác động của các yếu tố bất lợi [11;
23; 24]. Enzyme này còn có vai trò trong sự
dự trữ protein hay các hợp chất chứa nitơ
[19]. Ngoài ra, hai protein có chứa vùng bảo
thủ β-amylase (BAM7 và BAM8) hoạt động
như tác nhân điều hòa phiên mã kiểm soát
sự sinh trưởng của thân và sự phát triển của
Arabidopsis [20; 21].
Các β-amylase ở thực vật có chứa vùng
bảo thủ của họ glycosyl hydrolase 14 [4].
Cấu trúc không gian của phân tử protein có
các chuỗi β và các vòng với các trình tự đặc
trưng [18] cũng như có chứa nhiều xoắn α
trong cấu trúc bậc hai [13].
Trên quy mô hệ gene, 9 β-amylase đã
được phát hiện ở cây Arabidopsis thaliana
[5], 19 β-amylase đã được phát hiện ở cây
đậu tương (Glycine max) [2]. Phân tích cây
phả hệ cho thấy các β-amylase này được xếp
vào bốn nhóm khác nhau với cấu trúc khá
khác nhau [2]. Không phải tất cả các protein
này đều có mang các amino acid đặc trưng
và cũng không phải tất cả chúng đều có hoạt
tính enzyme β-amylase [5]. Cây sắn có chất
dự trữ trong củ chủ yếu là tinh bột, và các
50 Tạp chí Khoa học & Công nghệ số 4 (9) – 2017
gene liên quan đến sinh tổng hợp tinh bột
đã được nghiên cứu [15] trong khi mới chỉ
có những nghiên cứu về cấu trúc và hoạt
tính enzyme β-amylase [9; 14] nhưng chưa
có nghiên cứu toàn diện về họ gene này trên
quy mô hệ gene của cây sắn.
Nghiên cứu này hướng tới việc xác định
các gene mã hóa β-amylase trong hệ gene của
cây sắn, đồng thời phân tích các đặc điểm
của họ gene này bằng phương pháp tin sinh
học. Những kết quả bước đầu cung cấp các
thông tin khoa học có ý nghĩa về nghiên cứu
chức năng của các β-amylase trên cây lương
thực quan trọng này.
2.Vật liệu và phương pháp
nghiên cứu
2.1. Cơ sở dữ liệu về các trình tự hệ
gene và RNAseq ở cây sắn
Trình tự hệ gene của cây sắn được lấy từ
website phytozome (.
doe.gov/pz/portal.html#!info?alias=Org_
Mesculenta), nền tảng so sánh hệ gene
thực vật [7], phiên bản 1.0 thuộc dự án
PRJNA234389 của Bredeson et al. (2016) [1].
2.2.Xác định các gene thuộc họ
β-amylase ở cây sắn
Các β-amylase của cây Arabidopsis [5]
được dùng làm khuôn dò để tìm kiếm các
gene tương đồng trên dữ liệu nucleotide
của toàn hệ gene của cây sắn nhờ chương
trình TBLASTN.
2.3.Xây dựng cây phả hệ
Các protein β-amylase của cây Arabidopsis
và sắn được sắp dãy bằng MAFFT [12]. Cây
phả hệ được xây dựng bằng phần mềm
MEGA5 [22].
2.4.Phân tích các đặc điểm hóa–lý
Các đặc điểm vật lý, hóa học của các gene/
protein được phân tích nhờ các công cụ của
KHOA HỌC TỰ NHIÊN
ExPASy [6]. Cấu trúc CDS/intron được thiết
lập nhờ GSDS 2.0 [8].
giữa các protein β-amylase của cây sắn với
của cây Arabidopsis, giá trị dao động nằm
trong khoảng từ 31% đến 76% (bảng 2).
Khi so sánh các β-amylase cùng phân họ
kết quả cho thấy, mức độ tương tự giữa các
β-amylase phân họ I của hai loài thấp nhất
là 65% và cao nhất là 71%. Số liệu tương ứng
đối với các β-amylase phân họ II là 50% và
76%, phân họ III là 36% và 74%, phân họ IV
là 49% và 72%.
Như vậy, họ gene β-amylase của cây sắn
là họ đa gene (10 gene), kích thước tương
đồng với của cây Arabidopsis (9 gene) [5],
nhưng nhỏ hơn so với cây đậu tương (19
gene) [2].
3.Kết quả nghiên cứu và thảo luận
3.1. Xác định họ gene β-amylase ở
cây sắn
Nhờ sử dụng các protein β-amylase của
cây Arabidopsis làm khuôn dò trên toàn
hệ gene của cây sắn bằng chương trình
TBLASTN, tổng số 10 gene mã hóa cho
các β-amylase được tìm thấy trong hệ gene
của cây sắn (bảng 1). Phân tích trình tự
protein suy diễn cho thấy các gene đều mang
vùng bảo thủ của họ glycosyl hydrolase 14
(pfam 01373). Khi so sánh mức độ tương tự
Bảng 1. Các gene thuộc họ β-amylase của cây sắn và đặc điểm của chúng
Gene
MeBAM1
MeBAM2
MeBAM3
MeBAM4
MeBAM5
MeBAM6
MeBAM7
MeBAM8
MeBAM9
MeBAM10
Phân
nhóm
II
II
III
IV
IV
I
II
III
II
IV
Tên locus
Manes.02G006200
Manes.03G155800
Manes.03G191000
Manes.05G034800
Manes.05G034900
Manes.12G078500
Manes.15G047700
Manes.15G060100
Manes.15G171200
Manes.15G190300
Kích thước
gene (bp)
3158
2232
2180
6614
5676
4338
2553
7107
5823
34143
Chiều dài
protein (aa)
569
581
535
545
701
594
582
522
546
689
Khối lượng
protein (kD)
64,18
64,97
59,22
61,67
79,13
67,69
64,84
59,42
61,34
77,33
pI
8,59
5,67
6,04
5,69
5,63
6,04
6,05
8,92
8,7
5,44
Định khu
dưới tế bào
Lục lạp
Lục lạp
Lục lạp
Lục lạp
Tế bào chất
Tế bào chất
Tế bào chất
Ti thể
Tế bào chất
Nhân
Số
intron
3
3
2
8
9
7
3
9
3
9
Bảng 2. So sánh từng cặp protein β-amylase tương đồng của cây sắn và Arabidopsis. Giá trị biểu thị mức
độ tương đồng (%)
Gene
Sắn
MeBAM6
MeBAM1
MeBAM2
MeBAM7
MeBAM9
MeBAM3
MeBAM8
MeBAM4
MeBAM5
MeBAM10
Arabi
dopsis
Phân
nhóm
I
II
II
II
II
III
III
IV
IV
IV
AtBAM5 AtBAM6 AtBAM1 AtBAM3 AtBAM4 AtBAM9 AtBAM2 AtBAM7 AtBAM8
I
I
II
II
III
III
IV
IV
IV
71
37
49
51
48
34
42
54
50
41
65
35
46
47
48
34
42
55
53
44
48
50
76
76
60
40
44
50
47
40
48
50
62
58
72
41
45
52
48
42
41
38
43
42
46
38
74
42
38
42
31
34
37
36
38
57
36
34
35
35
47
37
49
50
50
35
42
72
71
57
44
35
43
44
45
34
38
68
68
49
39
37
38
40
41
37
36
59
52
69
Tạp chí Khoa học & Công nghệ số 4 (9) – 2017 51
KHOA HỌC TỰ NHIÊN
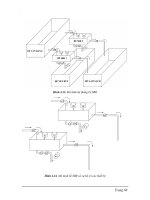
Hình 1. Bản đồ phân bố của các gene mã hóa β-amylase của cây sắn (chỉ thể hiện các NST có mang gene
mã hóa β-amylase)
3.2.Bản đồ gene, phân tích cây phả hệ
và phân loại các β-amylase
Các gene β-amylase phân bố trên 5 trong
tổng số 18 nhiễm sắc thể (NST) của cây sắn
(hình 1). Trong đó, NST số 15 mang nhiều
gene nhất (4 gene), NST số 3 và số 5 mang
2 gene, hai NST số 2 và số 12 mang một
gene. Kiểu phân bố này cũng bắt gặp ở cây
Arabidopsis cũng như ở cây đậu tương. Ở cây
Arabidopsis, các gene mã hóa cho β-amylase
Hình 2. Cây phả hệ được xây dựng từ các β-amylase
của cây sắn (Me) và cây Arabidopsis thaliana (At).
Cây phả hệ được xây dựng từ 19 trình tự protein của hai
loài sắn và Arabidopsis với các tham biến: thuật toán
Maximum Likelihood, mô hình Jones-Taylor-Thornton
(JTT), phương pháp Bootstrap với 1000 lần lặp lại, giá trị
bootstrap được thể hiện trên mỗi nhánh, thước tỷ lệ là số
amino acid thay thế trên một vị trí.
52 Tạp chí Khoa học & Công nghệ số 4 (9) – 2017
cũng chỉ phân bố trên 4 trong tổng số 5 NST
[5] còn ở cây đậu tương, chỉ có 12 trong tổng
số 20 NST có mang gene mã hóa β-amylase.
KHOA HỌC TỰ NHIÊN
Cây phả hệ được xây dựng từ 19 protein
β-amylase của hai loài sắn và Arabidopsis
sau khi chúng đã được sắp dãy được thể hiện
trong hình 2. Cây phả hệ chia làm bốn nhánh
tương ứng với bốn phân họ khác nhau được
kí hiệu từ I tới IV. Số lượng gene β-amylase
của cây sắn thuộc các phân họ I, II, III và IV
lần lượt là 1, 4, 2 và 3. Có một sự kiện nhân
gene trên quy mô hệ gene (Whole Geneome
Duplication, WGD) giữa MeBAM2 và
MeBAM7 được ghi nhận sau quá trình biệt
hóa giữa tổ tiên của Arabidopsis và sắn.
3.3. Đặc điểm các gene β-amylase ở
cây sắn
Các gene β-amylase của cây sắn có kích
thước và cấu trúc không giống nhau, chứa
từ 2.180 đến 3.4143 nucleotide (bảng 1).
Các gene β-amylase của cây sắn có số lượng
intron không giống nhau. Các gene phân họ
I có 7 intron, các gene phân họ II có 3 intron,
các gene phân họ III cùng có 2 hoặc 7 intron
và các gene phân họ IV có 8-9 intron (hình
3). Số lượng intron của các gene thuộc các
phân họ ở cây sắn tương đồng với ở cây đậu
tương đã được báo cáo [2].
Các gene β-amylase của cây sắn mã hóa
cho các protein có độ dài từ 522 tới 701 amino
acid, tương ứng với khối lượng phân tử từ
59,22 kD tới 79,13 kD. Các protein β-amylase
của cây sắn có điểm đẳng điện (pI) dao động
từ 5,44 tới 8,92 (bảng 1). Các protein phân
họ I và phân họ IV có tính axit (với giá trị pI
nhỏ hơn 7). Trong khi đó, các protein thuộc
phân họ II và phân họ III có tính kiềm hoặc
tính axit. Những đặc điểm vật lý này của các
β-amylase của cây sắn khá tương đồng với
các đặc điểm của các β-amylase đã được báo
cáo ở cây Arabidopsis [5].
Hình 3. Cấu trúc CDS (trình tự mã hóa)/intron của các gene β-amylase của cây sắn
Tạp chí Khoa học & Công nghệ số 4 (9) – 2017 53
KHOA HỌC TỰ NHIÊN
3.4. Các motif bảo thủ và cấu trúc của
các β-amylase ở cây sắn
Các β-amylase của cây sắn có chứa các
trình tự bộ phận bắt đầu với chuỗi beta
và tiếp tục nối với các vòng (β4/L4, β5/L5,
β6/L6, β7/L7, β8/L8). Các đoạn trình tự bộ
phận, ngoại trừ L3/H5, đều có chứa các
amino acid giữ vai trò quan trọng như trung
tâm xúc tác của enzyme, duy trì cấu trúc,
liên kết với cơ chất hay với các chất ức chế
[18]. Tuy nhiên, không phải tất cả các protein
trong họ đều có chứa đầy đủ các amino acid
giữ vai trò quan trọng trong chức năng của
enzyme, tương đồng với ở cây Arabidopsis
[5]. Riêng MeBAM5 và MeBAM10 thuộc
phân họ IV của cây sắn, ngoài vùng bảo
thủ của họ glycosyl hydrolase 14, còn chứa
đoạn amino acid giống với vùng bảo thủ của
tác nhân điều hòa phiên mã chống chịu với
Brassinazole (Brassinazole resistant, BZR1)
ở đầu amin (N-terminal). Kết quả này cũng
phù hợp với các kết quả phân tích cấu trúc
của hai gene BAM7 và BAM8 (phân họ IV)
của cây Arabidopsis [20].
4.Kết luận
Trong công trình này, chúng tôi đã xác
định và phân tích họ gene β-amylase ở cây
sắn bằng phương pháp tin sinh học với tổng
số 10 gene được phát hiện trong toàn hệ
Hình 4. Kết quả sắp dãy các protein β-amylase của cây sắn. Dấu đánh dấu các amino acid bảo thủ, các
vùng bảo thủ được đóng khung.
54 Tạp chí Khoa học & Công nghệ số 4 (9) – 2017
KHOA HỌC TỰ NHIÊN
gene. Các đặc điểm lý – hóa và cấu trúc của
tất cả các gene và protein suy diễn trong họ
cũng đã được xác định. Các motif bảo thủ
đã được tìm thấy trong hầu hết các protein
của họ β-amylase của cây sắn. Thông qua
phân tích cây phả hệ, chúng tôi đã phân loại
các β-amylase của cây sắn vào bốn phân họ
tương tự như ở các loài thực vật khác đã
được nghiên cứu. Bản đồ phân bố của các
gene trong họ này trên các nhiễm sắc thể đã
được xây dựng. Kết quả nghiên cứu này sẽ
mở đường cho việc tách dòng gene và phân
tích chi tiết chức năng của các gene trong họ
β-amylase ở cây sắn.
Lời cảm ơn
Công trình này được hoàn thành với sự
hỗ trợ kinh phí từ chương trình nghiên cứu
khoa học cơ bản của Trường Đại học Hùng
Vương, tỉnh Phú Thọ.
Tài liệu tham khảo
[1] Bredeson J. V., Lyons J. B., Prochnik S. E.,
Wu G. A., Ha C. M., Edsinger-Gonzales E.,
Grimwood J., Schmutz J., Rabbi I. Y., Egesi
C., Nauluvula P., Lebot V., Ndunguru J.,
Mkamilo G., Bart R.S., Setter T. L., Gleadow
R. M., Kulakow P., Ferguson M. E., Rounsley
S., Rokhsar, D. S. (2016). Sequencing wild
and cultivated cassava and related species
reveals extensive interspecific hybridization
and genetic diversity. Nat Biotechnol, 34(5),
562-570. doi:10.1038/nbt.3535
[2] Cao Phi Bằng, Trần Thị Thanh Huyền (2015).
Phân tích họ gen β-amylase ở cây đậu tương
(Glycine max). Tạp chí Sinh học, 37(1SE), 165176. doi:10.15625/0866-7160/v37n1se.6106
[3] Ceballos H., Okogbenin E., Pérez J. C., LópezValle L. A. B., Debouck D. (2010). Cassava.
In Root and tuber crops (pp. 53-96): Springer.
[4] Filiz E. (2014). In silico sequence analysis
and homology modeling of predicted
beta-amylase 7-like protein in Brachypodium distachyon L. 3(1), 7.
[5] Fulton D. C., Stettler M., Mettler T., Vaughan
C. K., Li J., Francisco P., Gil M., Reinhold H.,
Eicke S., Messerli G., Dorken G., Halliday
K., Smith A. M., Smith S. M., Zeeman S.
C. (2008). Beta-AMYLASE4, a noncatalytic
protein required for starch breakdown, acts
upstream of three active beta-amylases in
Arabidopsis chloroplasts. Plant Cell, 20(4),
1040-1058. doi:10.1105/tpc.107.056507
[6] Gasteiger E., Hoogland C., Gattiker A.,
Wilkins M. R., Appel R. D., Bairoch A.
(2005). Protein identification and analysis tools on the ExPASy server. In The
Proteomics Protocols Handbook (pp.571607): Springer.
[7] Goodstein D. M., Shu S., Howson R.,
Neupane R., Hayes R. D., Fazo J., Mitros T.,
Dirks W., Hellsten U., Putnam N., Rokhsar
D. S. (2012). Phytozome: a comparative
platform for green plant genomics. Nucleic
Acids Res, 40 (Database issue), D1178-1186.
doi:10.1093/nar/gkr944
[8] Guo A. Y., Zhu Q. H., Chen X., Luo J. C.
(2007). GSDS: a gene structure display
server. Yi Chuan, 29(8), 1023-1026.
[9] Hirata A., Adachi M., Utsumi S., Mikami
B. (2004). Engineering of the pH optimum
of Bacillus cereus beta-amylase: conversion
of the pH optimum from a bacterial type
to a higher-plant type. Biochemistry, 43(39),
12523-12531. doi:10.1021/bi049173h
[10] Howeler R., Lutaladio N., Thomas G. (2013).
Save and grow: Cassava. A guide to sustainable production intensification. Rome: FAO.
[11] Kaplan F., Guy C. L. (2004). Beta-Amylase
induction and the protective role of maltose
Tạp chí Khoa học & Công nghệ số 4 (9) – 2017 55
KHOA HỌC TỰ NHIÊN
during temperature shock. Plant Physiol,
135(3), 1674-1684. doi:10.1104/pp.104.040808
[12] Katoh K., Standley D. M. (2013). MAFFT
multiple sequence alignment software
version 7: Improvements in performance
and usability. Mol Biol Evol, 30(4), 772-780.
doi:10.1093/molbev/mst010
[13] Luo J. C., Wang S. C., Jian W. B., Chen C.
H., Tang J. L., Lee C. I. (2012). Formation
of amyloid fibrils from beta-amylase. FEBS
Lett, 586(6), 680-685. doi:10.1016/j.febslet.2012.01.062
[14] Mikami B., Degano M., Hehre E. J., Sacchettini J. C. (1994). Crystal structures of
soybean beta-amylase reacted with betamaltose and maltal: active site components
and their apparent roles in catalysis. Biochemistry, 33(25), 7779-7787.
[15] Munyikwa T. R. I., Langeveld S., Salehuzzaman S. N. I. M., Jacobsen E., Visser R.
G. F. (1997). Cassava starch biosynthesis:
new avenues for modifying starch quantity and quality. Euphytica, 96(1), 65-75.
doi:10.1023/A:1002935603412
[16] Oyefuga O. H., Adeyanju M. M., Adebawo
O. O., Agboola F. K. (2011). Purification
and some properties of β-amylase from the
nodes of sugar cane, Saccharium offinacium.
International Journal of Plant Physiology and
Biochemistry, 3(5), 117-124.
[17] Prochnik S., Marri P. R., Desany B., Rabinowicz P. D., Kodira C., Mohiuddin M.,
Rodriguez F., Fauquet C., Tohme J., Harkins
T., Rokhsar D. S., Rounsley S. (2012). The
Cassava Genome: Current Progress, Future
Directions. Tropical Plant Biology, 5(1),
88-94. doi:10.1007/s12042-011-9088-z
[18] Pujadas G., Ramirez F. M., Valero R., Palau
J. (1996). Evolution of beta-amylase: Patterns
of variation and conservation in subfamily
56 Tạp chí Khoa học & Công nghệ số 4 (9) – 2017
sequences in relation to parsimony mechanisms. Proteins, 25(4), 456-472. doi:10.1002/
prot.6
[19] Qi J. C., Zhang G. P., Zhou M. X. (2006).
Protein and hordein content in barley seeds
as affected by nitrogen level and their relationship to beta-amylase activity. Journal of
Cereal Science, 43(1), 102-107.
[20] Reinhold H., Soyk S., Simkova K., Hostettler C., Marafino J., Mainiero S., Vaughan
C. K., Monroe J. D., Zeeman S. C. (2011).
Beta-amylase-like proteins function as transcription factors in Arabidopsis, controlling
shoot growth and development. Plant Cell,
23(4), 1391-1403. doi:10.1105/tpc.110.081950
[21] Soyk S., Simkova K., Zurcher E., Luginbuhl
L., Brand L. H., Vaughan C. K., Wanke
D., Zeeman S. C. (2014). The Enzyme-Like
Domain of Arabidopsis Nuclear beta-Amylases Is Critical for DNA Sequence Recognition and Transcriptional Activation.
Plant Cell, 26(4), 1746-1763. doi:10.1105/
tpc.114.123703
[22] Tamura K., Peterson D., Peterson N.,
Stecher G., Nei M., Kumar S. (2011).
MEGA5: Molecular evolutionary genetics
analysis using maximum likelihood, evolutionary distance, and maximum parsimony
methods. Mol. Biol. Evol, 28(10), 2731-2739.
doi:10.1093/molbev/msr121
[23] Todaka D., Kanekatsu M. (2007). Analytical method for detection of beta-amylase
isozymes in dehydrated cucumber cotyledons by using two-dimensional polyacrylamide gel electrophoresis. Anal. Biochem,
365(2), 277-279. doi:10.1016/j.ab.2007.03.026
[24] Todaka D., Matsushima H., Morohashi Y.
(2000). Water stress enhances beta-amylase
activity in cucumber cotyledons. J. Exp. Bot,
51(345), 739-745.
KHOA HỌC TỰ NHIÊN
SUMMARY
Analysis of β-amylase encoded gene family in Cassava
(Manihot esculenta Crantz) by using bioinformatic methods
Cao Phi Bang
Faculty of Natural Sciences – Hung Vuong University
B
eta-amylases (β-amylase, EC 3.2.1.2) belong to the glycosyl hydrolase family 14
which function by hydrolyzing the 1,4-α-D-glycosidic linkages in starch-type polysaccharide substrates to remove successive maltose units from the non-reducing ends
of the chains. Using bioinformatic methods, a total of 10 genes encoding the β-amylase was identified in the whole geneome of cassava. These 10 genes distributed on
5 out of 18 chromosomes. Based on phylogenetic analysis, the cassava β-amylases
were classified into four subfamilies I, II, III and IV. All of 10 genes encoded discontinuously, their intron numbers differed up on the subfamily. The pairwise comparison
of predicted proteins from the same subfamily between the two species (cassava
and Arabidopsis) showed that they were similar. The pI values of soybean β-amylase
ranged from 5.44 to 8.92. All cassava β-amylases contained the conserved domain
of the glycosyl hydrolase family 14, and most of the proteins included amino acids
which are important for the enzymatic function. In addition, MeBAM5 and MeBAM10
proteins contained the amino acid sequence in N-terminal which was similar to the
transcription factors of the Brassinazole Resistant1 (BZR1) type.
Keywords: β-amylase, cassava, phylogenetic tree, gene characterization, bioinformatics,
gene identification.
Tạp chí Khoa học & Công nghệ số 4 (9) – 2017 57