Báo cáo thực hành KT di truyền PCR KHUẨN LẠC XÁC ĐỊNH DÒNG TẾ BÀO VI KHUẨN ,CHUYỂN GENE VÀO TẾ BÀO VI KHUẨN
Bạn đang xem bản rút gọn của tài liệu. Xem và tải ngay bản đầy đủ của tài liệu tại đây (1.8 MB, 24 trang )
Báo cáo thực hành
KĨ THUẬT DI TRUYỀN
BÀI 1
TẠO TẾ BÀO KHẢ NẠP VÀ TÁCH CHIẾT DNA PLASMID TỪ
TẾ BÀO VI KHUẨN
I. Nguyên tắc
- Các tế bào sẽ tiếp nhận các đoạn DNA một cách ngẫu nhiên. Khơng phải tất cả các lồi vi
khuẩn đều có khả năng tiếp nhận DNA. Ngay cả những lồi có khả năng này cũng chỉ có thể
tiếp nhận được DNA ở những pha sinh trưởng nhất định và trong những môi trường ni cấy
cụ thể. Các lồi Streptococcus pneumoniae và Bacillus subtilis tương đối dễ khả biến hơn,
trong khi E. coli phải mất đi hai loại enzyme exonuclease và phải được ni cấy trong mơi
trường có nồng độ cao của calcium chlodride để làm cho màng tế bào của nó có thể thấm
được DNA. Như vậy, những quá trình xử lý đặc biệt cũng được thực hiện nhằm gia tăng hiệu
quả chuyển gene ở vi khuẩn và làm chúng nhạy cảm hơn dưới tác động của cá chất hóa học
hay dịng điện trong kỹ thuật chuyển gene. Những tế bào vi khuẩn này được gọi là tế bào
khả nạp (competent cells).
- Quá trình chuyển gen đưa plasmid vào tế bào vi khuẩn là một bước quan trọng trong quy
trình tạo dịng giúp chọn lọc và nhân bản plasmid. Với những dòng vi khuẩn đã được sàng
lọc, tuyển chọn, việc nuôi cấy và tách chiết plasmid sẽ cho phép thu nhận một lượng lớn
plasmid để dùng cho các bước tiếp theo trong quy trình tạo dịng. Thơng thường, sinh khối vi
khuẩn sẽ được ly giải kiềm giúp giải phóng plasmid khỏi tế bào. Một bước xử lý acid hóa
nhanh bằng dung dịch kali acetate đậm đặc sẽ cho phép kết tủa protein và DNA nhiễm sắc
thể. DNA plasmid ở trạng thái siêu cuộn xoắn, vẫn ở trong dung dịch và có thể được giữ lại
trên cột silica spin. DNA plasmid được rửa bằng dung dịch ethanol và sau đó được rửa giải
trong nước hoặc đệm TE.
Hình 1. Quy trình tách chiết DNA plasmid từ sinh khối vi khuẩn dùng cột silica spin
II. Vật liệu:
- Vi khuẩn E. coli DH5-alpha
- Vi khuẩn BL21
- Môi trường LB lỏng
- Dung dịch CaCl2 100mM vô trùng
- Glycerol 100% vô trùng
2
- Bộ kit tách DNA plasmid
- Gel agarose 1.5% / 1 tấm
III. Quy trình thí nghiệm
1. Chuẩn bị tế bào khả nạp E. coli DH5-alpha và BL21
Cấy vi khuẩn vào ống nghiệm
chứa mơi trường LB khơng có
kháng sinh
Ni cấy lắc với tốc độ 150 rmp ở
370C trong thời gian 16-20h
Hút lần lượt 1ml dịch vi khuẩn sang
eppendorf
Ly tâm ở tốc độ 8000rpm trong 2
phút
Lặp lại các bước để thu nhận toàn
bộ sinh khối từ dịch vi khuẩn
Loại bỏ dịch nổi để thu sinh
khối vi khuẩn
Bổ sung 1ml dung dịch CaCl2 lạnh
Vortex để huyền phù sinh khối, ủ
eppendorf trong đá 30 phút
3
Ly tâm dịch vi khuẩn ở 8000 rpm
trong 2 phút
Loại bỏ dịch nổi để thu sinh
khối vi khuẩn
Bổ sung 500µl hỗn hợp dung dịch
CaCl2 và glycerol lạnh, vortex để
huyền phù sinh khối
Hút chuyển lần lượt 100µl dung dịch vi
khuẩn vào các eppendporf lạnh
vô trùng
Bảo quản các ống vi khuẩn ở nhiệt độ
-80oC
2. Quy trình thu nhận sinh khối vi khuẩn tách chiết DNA plasmid từ sinh khối vi
khuẩn bằng bộ kit
10 mL dung dịch vi khuẩn
chứa plasmid mục tiêu (pha
lag)
Hút 1 mL dịch nuôi cấy
Ly tâm ở 11000 rpm trong 5
phút
4
Loại bỏ dịch nổi
Thu cặn tế bào
Cho 250 mL PL1 buffer + 10 µl RNase A vào
tube chứa 1.5 mL cặn tế bào
Vortex
Thêm 250 µl PL2 buffer
Đảo tube 6-8 lần
Ủ ở nhiệt độ phịng khoảng
5 phút
Thêm 350 µl PL3 buffer
Đảo tube 6-8 lần
Ly tâm ở 11000 rpm trong 5
phút
Chuyển tối đa 600 µl sang cột
silica
5
Ly tâm ở 11000 rpm trong 2
phút
Loại bỏ chất lỏng bên
dưới
Giữ lại cột
Ly tâm ở 11000 rpm trong 2
phút
Loại bỏ chất lỏng bên
dưới
Giữ lại cột
Đặt cột vào tube cũ
Thêm 500 µl WB1 buffer
Ly tâm ở 11000 rpm
trong 2 phút
Loại bỏ chất lỏng bên
dưới
Giữ lại cột
Đặt cột vào tube cũ
Thêm 500 µl WB2 buffer
Ly tâm ở 11000 rpm trong 2
phút
Loại bỏ chất lỏng
bên dưới
Giữ lại cột
6
Ly tâm ở 11000 rpm trong
30 giây
Chuyển cột sang tube mới
Thêm 50 µl EB buffer
Ủ ở nhiệt độ phịng khoảng
1 phút
Ly tâm ở 11000 rpm trong 1
phút
Thu dịch chứa DNA
Đo OD
Bảo quản DNA plasmid ở -20ºC
7
IV. Kết quả thí nghiệm:
Hình 2. Kết quả phân tích plasmid bằng phương pháp đo mật
độ quang
V. Thảo luận kết quả:
- Sau khi tiến hành tách chiết DNA plasmid, để xác định nồng độ của DNA trong dung dịch,
nhóm thực hiện phân tích plasmid bằng phương pháp đo mật độ quang bằng máy quang phổ
nanodrop. Kết quả thu được DNA plasmid với nồng độ rất thấp lần lượt là 46 và 55 µg/mL.
- Nồng độ DNA plasmid thấp như trên có thể do lượng DNA và buffer trong thí nghiệm chưa
thật sự chính xác (do đầu típ khơng khớp với micropipette)…
8
BÀI 2
CẮT DNA BẰNG ENZYME CẮT GIỚI HẠN (RESTRICTION
ENZYME)
I. Nguyên tắc
Enzyme cắt giới hạn (restriction enzyme-RE) là enzyme cắt DNA tại vị trí nhận biết đặc hiệu
để tạo đầu bằng (blunt end) hoặc đầu dính (sticky end). Trong phương pháp tạo dòng truyền
thống, RE được sử dụng để cắt các DNA tạo các đầu tương hợp giúp kết hợp chúng với nhau
tạo DNA tái tổ hợp. Nguồn DNA sử dụng trong tạo dịng rất đa dạng, có thể là DNA tách
chiết từ tế bào vi khuẩn hoặc từ quần thể vi sinh vật, hoặc là dòng DNA cần được chuyển từ
vector này sang vector khác (subcloning). RE cũng được sử dụng để tạo các đầu tương hợp
cho sản phẩm PCR. Trong tất cả các trường hợp, một hay nhiều RE được sử dụng để cắt
DNA giúp chèn có định hướng hoặc khơng định hướng vào plasmid tương thích.
DNA bộ gene (genome), từ bất kỳ nguồn nào, cũng thường được cắt với RE tại vị trí nhận
biết đặc trưng gồm 6-8 base liên tiếp, là dạng vị trí nhận biết với tần suất trong genome ít
hơn so với vị trí nhận biết 4 base, dẫn đến tạo các phân đoạn DNA có kích thước lớn. Kích
thước của các đoạn chèn mong muốn của thư viện dòng nhân bản sẽ quyết định loại enzyme
và điều kiện của phản ứng cắt. Thông thường, một chuỗi các độ pha loãng của enzyme được
lựa chọn thường được sử dụng để phân cắt nguồn vật liệu khởi đầu, sau đó kích thước các
đoạn chèn các đoạn chèn mong muốn được phân tách bằng điện di trên gel agarose. Phương
pháp chuẩn bị này tạo các phần đoạn DNA có kích thước mong muốn với các đầu tương
thích với các vector đã lựa chọn.
Thơng thường, quy trình subcloning yêu cầu sử dụng 1-2 RE cắt ngay bên ngoài đoạn chèn
mà khơng cắt trong chính đoạn chèn. Các RE, có một vị trí nhận biết trong vùng đa điểm tạo
dòng (multi cloning sites-MCS), thường được chọn sử dụng khi chúng khơng cắt vị trí khác
trong DNA vector và thường tạo ra hai phân đoạn dễ tách biệt. Gene mục tiêu được tạo dòng
vào một vector với mục tiêu nâng cao hiệu quả biểu hiện thành protein hoặc giúp sàng lọc
gene. Trong những trường hợp này, gene mục tiêu cần phải được gắn chèn vào đúng hướng
và năm trong khung đọc với promoter phiên mã.
Phản ứng tổng hợp dây chuyền nhờ enzyme polymerase (Polymerase Chain Reaction - PCR)
thường được sử dụng để khuếch đại gene hay phân đoạn DNA của gene mục tiêu, từ bất kỳ
nguồn DNA nào, để tạo dịng. Để tạo các đầu tương thích, thường trình tự nhận biết của RE
sẽ được thêm vào đầu 5' của cả hai mồi PCR. Hơn nữa, để tạo hiệu quả gắn kết và cắt của
RE, một số gốc base cũng sẽ được thêm vào phía trước và phía sau điểm nhận biết ở đầu 5'.
Khi chọn vị trí cắt giới hạn để thêm vào trình tự mồi, điều quan trọng là xác định vị trí cắt
phải tương hợp với vector, chiều của các đoạn chèn và vị tri cắt không xảy ra trong gene
hoặc trong phân đoạn DNA.
9
II. Vật liệu và phương pháp
1. Vật liệu
-
Plasmid pET11a: 10µl
-
EcoRI: 10µl
-
BamHI: 10µl
-
Buffer 10X: 10µl
-
Gel agarose 1.5%: 1 tấm
-
DNA marker 1kb: 5µl
2. Phương pháp
-
Thực hiện phản ứng cắt plasmid DNA với enzyme BamHI
-
Thực hiện phản ứng cắt plasmid DNA với 2 enzyme là EcoRI và BamHI
III. Quy trình thí nghiệm
1. Thực hiện phản ứng cắt plasmid DNA với enzyme BamHI
15µl DNA
2µl Buffer
1µl Enzyme BamHI
Chuẩnbị
bị eppendorf
eppendorf
Chuẩn
Trộn
bằng
Trộn nhẹ bằng
micropipette
micropipette
Ủ ở 370C trong
60 phút
10
2. Thực hiện phản ứng cắt plasmid DNA với 2 enzyme là EcoRI và BamHI
16µl DNA
2µl Buffer
1µl Enzyme BamHI và 1µl
Enzyme EcoRI
v
va
Chuẩn bị eppendorf
Trộn nhẹ bằng
micropipette
Ủ ở 370C trong 60
phút
3. Điện di kiểm tra kết quả cắt DNA bằng enzyme cắt giới hạn
Nạp thang
chuần
Chuẩn bị bản gel
agarose 1.5%
Nạp mẫu phản
ứng cắt
14,5µl mẫu khơng cắt và 3µl
loading dye
20µl mẫu cắt bằng 1 enzyme
và 4µl loading dye
20µl mẫu cắt bằng 2 enzyme
và 4µl loading dye
Điện di ở 100 voltage
trong 60 phút
Quan sát và so sánh
11
IV. Kết quả thí nghiệm:
Sau khi điện di bản gel agarose 1.5% ở 100 voltage trong 60 phút , ta thu được kết quả như
hình bên dưới
Hình 3. Kết quả phân tích plasmid bằng phương pháp điện di trên gel agarose
(L: Ladder 1kb; 1-2: DNA plasmid không cắt; 3-4: DNA plasmid được cắt bởi BamHI; 5-6:
DNA plasmid được cắt bởi 2 enzyme EcoRI + BamHI và EcoRI + PstI; 7-8: DNA plasmid
khơng cắt của nhóm sáng)
V. Thảo luận kết quả
- Sau khi có được kết quả điện di, nhận thấy các vạch điện di xuất hiện không rõ ràng, không
phân đoạn tách biệt. Ở DNA marker dùng làm mẫu nên xuất hiện rõ ràng hơn sản phẩm cắt
plasmid của mẫu thí nghiệm, điều này khó xác định được sự phân cắt của hai phản ứng cắt
enzyme cắt giới hạn. Vì thế không so sánh được khả năng cắt plasmid bằng RE so với
plasmid chưa được cắt.
- Sau khi điện di, các vạch điện di xuất hiện khơng rõ ràng có thể là do tỉ lệ đổ bản gel
agarose không phù hợp ảnh hưởng đến sự di chuyển của các đoạn DNA trong mơi trường
diện di, hay cũng có thể do dung dịch điện di không đạt chuẩn ảnh hưởng đến kết quả.
VI. Trả lời câu hỏi
Vì sao phản ứng cắt plasmid DNA với một enzyme giới hạn lại sử dụng enzyme BamHI mà
khơng sử dụng enzyme EcoRI?
Trả lời: Bởi vì vị trí nhận biết để phân cắt của enzyme BamHI có vị trí là 319bp nằm gần
hơn nhiều so với vị trí nhận biết của enzyme EcoRI (5675bp) vì thế hiệu quả cắt của enzyme
BamHI trên plasmid sẽ dễ dàng hơn.
12
BÀI 3
CHUYỂN GENE VÀO TẾ BÀO VI KHUẨN BẰNG PHƯƠNG PHÁP
SHOCK NHIỆT VÀ TÁCH CHIẾT LẠI DNA PLASMID
I. Nguyên tắc:
Chuyển gen là quá trình đưa DNA ngoại lai vào tế bào sinh vật. Quá trình chuyển
plasmid vào tế bào vi khuẩn (q trình biến nạp) khơng chỉ quan trọng trong nghiên
cứu mà còn trong nhân bản và lưu trữ plasmid. Bởi vì điều này, hầu như tất cả các
plasmid, thậm chí các plasmid được thiết kế cho ứng dụng ở thực vật và động vật,
cũng đều mang vùng khởi đầu sao chép (ori) và vùng gene kháng kháng sinh dùng
như một marker để chọn lọc dịng vi khuẩn. Có nhiều biến đổi di truyền được thực
hiện để tạo các chủng vi khuẩn có thể chuyển gene vào dễ dàng hơn cũng như giúp
duy trì plasmid mà khơng làm thay đổi plasmid.
Hình 4. Plasmid pET11a
II. Vật liệu:
Plasmid pET11a: 2µl
Vi khuẩn khả nạp E. coli DH5-alpha và BL21: 100µl
Mơi trường LB agar: 50ml
Đĩa petri chứa môi trường LB ager bổ sung kháng sinh (ampicilin)
13
III. Quy trình thí nghiệm
1. Quy trình chuyển gene vào tế bào vi khuẩn khả nạp bằng phương pháp shock
nhiệt
Giải đông ống vi khuẩn khả nạp
vào hộp đá trong 5-10 phút
Hút bổ sung 5µl của plasmid vào vi
khuẩn khả nạp đã giải đông. Trộn nhẹ
hỗn hợp bằng micropipette
Đặt ống eppendorf chứa hỗn hợp biến
nạp trong chậu nước đá trong 5 phút
Chuyển eppendorf vào bể ổn nhiệt ở
42oC trong 42 giây
Đặt eppendorf lại vào chậu nước đá
trong 5 phút
Bổ sung 1ml môi trường LB
Ủ trên máy lắc 37oC trong 45 phút
Cấy trải toàn bộ dung dịch vi khuẩn
vào đĩa petri chứa mơi trường LB agar
có bổ sung kháng sinh
14
Nuôi ủ đĩa petri ở 37oC qua đêm
Chọn 10 khuẩn lạc mọc rời trên đĩa
petri
Thu được chủng vi khuẩn kháng kháng
sinh
IV. Kết quả thí nghiệm
Hình 5. Kết quả sàng lọc thể biến nạp dựa trên khả năng kháng kháng sinh
1-Vi khuẩn BL21 đã chuyển gen; 2- Vi khuẩn DH5-alpha không chuyển gen (đối chứng)
15
Hình 6. Kết quả phân tích plasmid bằng phương pháp đo mật độ quang (lần 2)
- Thực hiện cắt DNA bằng enzyme cắt giới hạn với nồng độ DNA plasmid cao hơn (68
µg/ml)
L
Uncut
Cut1
Cut2
Hình 7. Kết quả phân tích plasmid bằng phương pháp điện di trên gel agarose (lần 2)
(L: Ladder 1kb; Uncut: DNA plasmid không cắt với nồng độ 68 µg/ml; Cut1: DNA plasmid
được cắt bởi BamHI; Cut2: DNA plasmid được cắt bởi 2 enzyme EcoRI + BamHI)
- Thảo luận kết quả:
+ Sau khi thực hiện cắt DNA bằng enzyme cắt giới hạn với nồng độ DNA plasmid cao hơn
hơn (68 µg/ml), tuy nhiên kết quả đạt được khơng như mong muốn, trên bản gel không xuất
hiện bất cứ band nào ngồi các band trên thang chuẩn.
+ Ngun nhân có thể do nồng độ DNA plasmid này quá thấp nên khi cắt DNA không thu
được kết quả.
16
V. Thảo luận kết quả
1. Kết quả sàng lọc thể biến nạp dựa trên khả năng kháng kháng sinh
Xét về khả năng sinh trưởng của vi sinh vật, chủng DH5-alpha sinh trưởng phát triển mạnh
hơn nhiều so với chủng BL21, cho nên với lượng 100 µl ampicilin 50mM, ta thấy:
- Đĩa cấy vi khuẩn BL21 đã chuyển gen kháng kháng sinh không xuất hiện khuẩn lạc (không
phát triển)
- Mặc khác, chủng DH5-alpha phát triển mạnh mẽ trên đĩa môi trường LB bổ sung kháng
sinh ampicilin
Do vậy, không thể đánh giá kết quả sàng lọc thể biến nạp.
2. Kết quả phân tích DNA plasmid
+ Sau khi thực hiện cắt DNA bằng enzyme cắt giới hạn với nồng độ DNA plasmid cao hơn
hơn (68 µg/ml), tuy nhiên kết quả đạt được khơng như mong muốn, trên bản gel không xuất
hiện bất cứ band nào ngoài các band trên thang chuẩn.
+ Nguyên nhân có thể do nồng độ DNA plasmid này quá thấp nên khi cắt DNA không thu
được kết quả.
17
BÀI 4
PHƯƠNG PHÁP PCR KHUẨN LẠC XÁC ĐỊNH DÒNG TẾ BÀO VI
KHUẨN MANG GENE MỤC TIÊU
I.
Nguyên tắc:
Phương pháp PCR khuẩn lạc (colony PCR) là phương pháp rất thuận lợi và hiệu quả trong
việc sàng lọc số lượng lớn các dòng vi khuẩn để xác định sự hiện diện hay vắng mặt của
plasmid mang gene mục tiêu trong quá trình tạo dịng. Các dong vi khuẩn chuyển gene có
thể được ly giải trong nước hoặc dung dịch đệm trong một bước xử lý nhiệt trước khi bổ
sung vào hỗn hợp phản ứng PCR hoặc cũng có thể bổ sung trực tiếp vào phản ứng và được
ly giải trong giai đoạn khởi đầu của chu kỳ nhiệt. Bước xử lý nhiệt trong giai đoạn khởi đầu
cho phêp giải phóng plasmid DNA từ tế bào vi khuẩn làm nguyên liệu (mạch khuôn) cho
phản ứng PCR.
Trong phản ứng PCR, với một cặp mồi đặc hiệu cho gene mục tiêu cho phép xác định sự
hiện diện của gene mục tiêu trong cấu trúc của plasmid. Ngoài ra, với các mồi chuyên biệt
được thiết kế cho các vùng gene trên khung vector, bên cạnh gene mục tiêu, cho phép xác
định chiều và kích thước của gene mục tiêu. Trong tất cả các trường hợp, sản phẩm của phản
ứng PCR được xác định bằng điện di trên gel agarose.
Hình 8. Quy trình PCR khuẩn lạc
18
II.
Vật liệu và phương pháp:
1. Vật liệu:
- Các chủng vi khuẩn được sàng lọc
- Mồi xuôi 20M: 12l
- Mồi ngược 20M: 12l
- MyTaq mix 2X: 150l
2. Phương pháp:
- Phản ứng PCR.
- Phân tích kết quả bằng điện di gel agarose.
III. QUY TRÌNH THÍ NGHIỆM:
1. Chuẩn bị phản ứng colony PCR:
(*) Hút các thành phẩn phản ứng theo bảng sau:
Các thànhChuẩn
phầnbị eppendorf sạch
Thể tích
H2O
10.5 l
MyTaq mix 2X
l
Hút các thành phần phản ứng PCR vào 12.5
eppendorf
(*)
Mồi xi 20M
1 l
Mồi ngược 20M
1 l
Hồ tan khuẩn lạc vào dung dịch phản ứng bằng đầu típ vơ trùng
Vortex và spindown nhanh các ống phản ứng
2. Quy trình nhiệt của phản ứng colony PCR: được cài đặt theo bảng sau:
Các
nhiệt
bước
950C
950C
620C
720C
720C
40C
Thời gian
Số lần lặp lại
10 phút
30 giây
30 giây
30 giây
10 phút
1 lần
30 lần
1 lần
19
3. Điện di kiểm tra kết quả phản ứng colony PCR:
Chuẩn bị bản gel agarose 1,5%
IV.
Trộn 25l sản phẩm PCR vào 6,25l gel loading buffer (GLB)
Nạp mẫu vào gel
Điện di sản phẩm cắt ở 100 voltage trong 30 phút
Quan sát xác định band sản phẩm PCR
So sánh với marker để xác định kích thước
Kết quả thí nghiệm
Sau khi tiến hành thí nghiệm ta có được kết quả như sau:
Hình 9. Kết quả phân tích sản phẩm PCR bằng phương pháp điện di trên gel agarose
(L: Ladder 100bp – 1013bp, 1-10: Sản phẩm PCR khuẩn lạc vi khuẩn chứa gen mục tiêu, 1112: Đối chứng âm - dương)
20
V. Thảo luận
*Sau khi điện di sử dụng thuốc nhuộm gel safeview:
- Trong thời gian chờ gel đơng thì safeview đã bị mất màu.
- Qua nhiều lần sử dụng safeview thì khơng hiển thị được các band sản phẩm PCR.
Nên có thể thuốc nhuộm safeview đã bị hỏng hoặc hết hạn sử dụng. Và các thao tác chưa
đúng nên ảnh hưởng đến kết quả.
*Nhóm thực hành đã quyết định chọn thuốc nhuộm Ethidium bromide (EtBr) để thay thế
cho safeview:
- Cách thực hiện:
Điện di trên gel agarose 1.5%
Dùng sản phẩm điện di ngâm vào EtBr 15 phút
Quan sát xác định band sản phẩm PCR trong hộp đèn soi UV
So sánh với marker để xác định kích thước
-
Sau khi thay đổi thuốc nhuộm vẫn không thể thấy được các band của sản phẩm PCR.
Có thể do thao tác thực hiện cịn nhiều sai sót, hố chất khơng đảm bảo chất lượng, mồi
q dài (không đặc hiệu) nên kết quả không thể xác định được các band sản phẩm PCR
và xác định được kết qủa chính xác.
Bổ sung kết quả thực hiện lần 2
Do PCR khuẩn lạc không đạt được kết quả mong muốn, tiếp tục tiến hành thực hiện lại thí
nghiệm lần 2 với một số thay đổi:
+ Giữ nguyên thể tích của các thành phần: MyTaq mix và nước, thay đổi thể tích của mồi
xi và mồi ngược:
Các thành phần
H2O
MyTaq mix 2X
Mồi xi 20M
Mồi ngược 20M
Thể tích
10.5 l
12.5 l
0.25 l
0.25 l
+ Giữ nguyên quy trình nhiệt, thay đổi nhiệt độ bắt cặp:
Các
bước
Thời gian
Số lần lặp lại
21
nhiệt
950C
950C
520C
720C
720C
40C
10 phút
30 giây
30 giây
30 giây
10 phút
1 lần
30 lần
1 lần
Kết quả điện di PCR khuẩn lạc lần 2
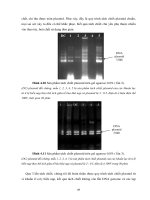
Hình 10. Kết quả phân tích sản phẩm PCR bằng phương pháp điện di trên gel agarose
(L: Ladder có kích thước từ 100 bp – 1013bp; 1-10: Sản phẩm PCR khuẩn lạc vi khuẩn
chứa gen mục tiêu; 11-12 : Đối chứng dương : Sinh khối sau khi ly tâm dịch vi khuẩn được
cấy trong môi trường LB lỏng chứa Amp)
Thảo luận kết quả
- Sau khi thay đổi một số điều kiện của phản ứng PCR colony, nhìn vào bản gel sau khi điện
di ta thấy ở giếng số 4 và số 10 xuất hiện các band, điều chứng tỏ gen mục tiêu đã được chèn
thành công vào plasmid ở các khuẩn lạc này. Ước tính kích thước plasmid chứa đoạn gen ở
giếng số 10 khoảng 1000bp.
- Ở các giếng còn lại (ngoại trừ ladder) không xuất hiện band nào, kết luận các plasmid
không được chèn thành công gen mục tiêu ở các khuẩn lạc này.
22