Nghiên cứu đa dạng nguồn gen di truyền quần thể thông lá dẹt (pinus krempfii lecomte) ở tây nguyên loài đặc hữu của việt nam
Bạn đang xem bản rút gọn của tài liệu. Xem và tải ngay bản đầy đủ của tài liệu tại đây (1.87 MB, 71 trang )
1
BỘ GIÁO DỤC VÀ ĐÀO TẠO
VIỆN HÀN LÂM KHOA HỌC
VÀ CÔNG NGHỆ VIỆT NAM
VIỆN SINH THÁI VÀ TÀI NGUYÊN SINH VẬT
TRẦN THỊ LIỄU
“NGHIÊN CỨU ĐA DẠNG NGUỒN GEN DI TRUYỀN QUẦN
THỂ THÔNG LÁ DẸT (Pinus krempfii Lecomte) Ở TÂY
NGUYÊN – LOÀI ĐẶC HỮU CỦA VIỆT NAM”
LUẬN VĂN THẠC SĨ SINH HỌC
HÀ NỘI - NĂM 2014
2
BỘ GIÁO DỤC VÀ ĐÀO TẠO
VIỆN HÀN LÂM KHOA HỌC
VÀ CÔNG NGHỆ VIỆT NAM
VIỆN SINH THÁI VÀ TÀI NGUYÊN SINH VẬT
TRẦN THỊ LIỄU
Đề tài :
“NGHIÊN CỨU ĐA DẠNG NGUỒN GEN DI TRUYỀN QUẦN
THỂ THÔNG LÁ DẸT (Pinus krempfii Lecomte) Ở TÂY
NGUYÊN – LOÀI ĐẶC HỮU CỦA VIỆT NAM”
Chuyên ngành: Sinh học thực nghiệm
Mã số:
60 42 01 14
LUẬN VĂN THẠC SĨ SINH HỌC
Ngƣời hƣớng dẫn: PGS. TS. Đinh Thị Phòng
Bảo tàng Thiên nhiên Việt Nam
.TSGS. TS. Lê Xuân
HÀ NỘI, NĂM 2014
3
MỞ ĐẦU
Tây Nguyên là một trong những vùng giàu loài lá kim nhất Việt Nam. Hầu
hết những loài lá kim ở Tây Nguyên đều là những loài có giá trị khoa học và kinh tế
cao. Nhiều loài đang đứng trước nguy cơ bị đe dọa tuyệt chủng, trong đó có loài
Thông lá dẹt (Pinus krempfii Lecomte) hay còn gọi là Thông hai lá dẹt, Thông Sri,
Thông Sré, là loài đặc hữu hẹp của Việt Nam [4]. Đây là nguồn gen quý và độc đáo
của với lá hình dải mác không hình kim như các loài Thông khác [32], [33]. Gỗ
Thông lá dẹt mềm, ít nhựa, màu từ trắng đến vàng nhạt, nhẹ, có nhiều đặc tính kỹ
thuật tốt. Hiện nay các rừng Thông lá dẹt đang bị đe dọa nghiêm trọng do tình trạng
phá rừng làm nương rẫy, nhiều cây bị mất môi trường sinh sống tối ưu nên chết rụi,
một số cây quá già cũng tự đổ gãy dẫn đến suy giảm số lượng loài nghiêm trọng.
Tái sinh tự nhiên hầu như chỉ hạn chế ở giai đoạn cây mầm, lại gặp chủ yếu ở nơi có
khoảng trống, ven đường. Mặt khác lại thiếu vắng các cây tái sinh ở tuổi trung gian,
nên khó đủ sức thay thế những cánh rừng Thông lá dẹt cổ thụ đang tồn tại [5], [6].
Theo Quỹ Bảo tồn Thiên nhiên quốc tế (IUCN) 2013 Thông lá dẹt được xếp vào
bậc sắp bị tuyệt chủng VU A2c, B1ab (iii) [65]. Vì vậy, việc bảo tồn hiệu quả
nguồn gen Thông lá dẹt là nhiệm vụ cấp bách đặt ra cho các nhà nghiên cứu. Tuy
nhiên, các nghiên cứu trước đây mới chỉ tập trung vào việc phân loại dựa trên đặc
điểm hình thái và nơi phân bố, còn các nghiên cứu về đa dạng di truyền nguồn gen
vẫn rất hạn chế và mới chỉ tập trung cho một số loài [7], [8], [46]. Đặc biệt các dẫn
liệu về đa dạng nguồn gen di truyền, các trình tự nucleotide đặc trưng cho loài
Thông lá dẹt hầu như chưa được nghiên cứu.
Với sự phát triển mạnh mẽ của công nghệ sinh học hiện đại, nhiều loại chỉ
thị phân tử đã được sử dụng để đánh giá đa dạng di truyền nguồn gen làm cơ sở cho
nghiên cứu bảo tồn và tái tạo nguồn gen ở đối tượng sinh vật nói chung và ở các
loài cây lá kim nói riêng [7], [10], [29], [34], [56], [68]. Trong các loại chỉ thị thì
chỉ thị ISSR (Internal Sequence Simple Repeat) và SSR (Sequence Simple Repeat)
đang được ứng dụng rộng rãi và có hiệu quả trong việc đánh giá đa dạng di truyền ở
cả mức độ quần thể và loài. Hơn nữa, phân tích phân tử và nhận dạng vùng gen đặc
4
trưng cũng được sử dụng rộng rãi trên thế giới và cả ở Việt Nam để đánh giá các mô
hình đa dạng di truyền ở thực vật và kỹ thuật này có lợi thế tiềm năng cho việc điều
tra thực vật quý hiếm. Vì thế đến nay đã có một số công trình nghiên cứu công bố
về hiệu quả cao của phân tích phân tử trong nghiên cứu đa dạng di truyền và xác
định trình tự nucletide vùng gen đặc trưng cho một số loài lá kim của Việt Nam [2],
[3], [7]. Điển hình là công bố của Vũ Đình Duy và cộng sự (2010) về việc sử dụng
chỉ thị ISSR và SSR đánh giá đa dạng di truyền của 4 loài lá kim là Pơ mu
(Fokienia hodginsii), Sa mộc dầu (Cunninghamia lanceolata var. konishii), Hoàng
đàn hữu liên (Cupressus tonkinesis) Thủy tùng (Glytostrobus pensilis) và giải mã
trình tự vùng gen 18S đã chỉ ra hai loài Pơ mu và Sa mộc dầu có mức độ suy giảm
cao hơn loài Thủy tùng và Hoàng đàn hữu liên. Kết quả giải mã trình tự vùng gen
18S còn cho phép giải quyết vấn đề tồn tại về taxon của Sa mộc dầu (Cunninghamia
lanceolata var. konishii). Tương tự, Đinh Thị Phòng và cộng sự (2009), Vũ Thi Thu
Hiền và cộng sự (2009) cũng đã sử dụng chỉ thị thị RAPD, ISSR và cpSSR để
nghiên cứu mối quan hệ di truyền loài Pơ mu, Bách xanh phục vụ cho cho công tác
bảo tồn. Hiện nay trong ngân hàng Genbank (2014) đã lưu giữ trên 884.000 trình tự
nucleotide cho các loài lá kim (conifers), tập trung vào hai vùng gen chính là nhân
(ITS) và vùng gen lục lạp (cpDNA), làm cơ sở cho xác định trình tự nucleotide đặc
trưng cho loài Thông lá dẹt.
Xuất phát từ các cơ sở khoa học trên đây, chúng tôi tiến hành đề tài “Nghiên
cứu đa dạng nguồn gen di truyền quần thể Thông lá dẹt tự nhiên (Pinus
krempfii Lecomte) ở Tây Nguyên - loài đặc hữu của Việt Nam” với các mục tiêu
và nội dung nghiên cứu sau:
- Xác định mức độ đa dạng nguồn gen di truyền cho 4 quần thể Thông lá dẹt
tự nhiên thu tại các tỉnh ở Tây Nguyên bằng chỉ thị SSR và ISSR.
- Xác định trình tự nucleotide đặc trưng tại 3 vùng gen (2 vùng gen lục lạp và
1 vùng gen nhân) cho loài Thông lá dẹt ở Tây Nguyên.
5
CHƢƠNG I. TỔNG QUAN TÀI LIỆU
1.1. Giới thiệu tổng quát về loài Thông lá dẹt
1.1.1. Vị trí phân loại
Thông lá dẹt (Pinus krempfii Lecomte) còn gọi là Thông hai lá dẹt hay
Thông Sri, Thông Sré, là loài đặc hữu của Việt Nam, phân bố ở các tỉnh Khánh
Hòa, Lâm Đồng, Đắc Lắc và Ninh Thuận. Theo phân loại khoa học, Thông lá dẹt
thuộc:
Giới (regnum): Plantae
Ngành (phylum): Pinophyta
Lớp (class): Pinopsida
Bộ (order): Pinales
Họ (familia): Pinaceae
Chi (genus): Pinus
1.1.2. Một số đặc điểm sinh học chính và giá trị bảo tồn loài Thông lá dẹt
Theo đánh giá của các nhà khoa học trong và ngoài nước, Thông lá dẹt là
loài cổ sinh vật hiếm hoi còn sót lại cho đến ngày nay, với chiều cao lên đến 30 m,
đường kính có thể đạt 1,5 - 1,6 m, đôi khi tới 2 m [47]. Tán của cây thường khá
rộng, dày, sẫm màu và có hình rẻ quạt. Đoạn thân dưới cành lớn, hầu như không có
cành nhánh, tròn đều và mọc thẳng lên (Hình 1.1). Nón xuất hiện vào tháng 4 - 5 và
tồn tại trên cây trong một thời gian dài. Hạt chín vào tháng 7- 10, màu nâu nhạt và
có cánh trắng. Khi chín, hạt có thể phát tán trong một phạm vi tương đối rộng. Cây
mầm thường có khoảng 10 - 13 lá mầm đầu tiên có hình xoắn cong về một hướng
như lưỡi liềm, lá dài khoảng 2 - 3 cm, sau đến là các lá nhỏ mọc quanh thân, dài 1,5
- 2,5 cm. Khi cây còn non, lá dài và rộng bản (dài 10 - 15 cm) xếp như hai lưỡi kéo
ở phần đầu cành. Khi cây trưởng thành, lá nhỏ và ngắn lại (dài 4 - 5 cm), màu sẫm,
mọc tập trung ở đầu cành, làm cho tán cây thông già trở nên dày và sẫm màu hơn.
Đặc điểm đặc trưng nhất là lá hình dải mác nhọn đầu, dẹt [32], [33].
6
B
A
C
Hình 1.1. Hình ảnh cây Thông lá dẹt ở Lâm Đồng (A: cây trưởng thành; B: nón
quả; C: cây tái sinh) (ảnh Nguyễn Tiến Hiệp)
Gỗ Thông lá dẹt mềm, ít nhựa, màu từ trắng đến vàng nhạt, nhẹ, có nhiều đặc
tính kỹ thuật tốt có thể sử dụng trong công nghiệp và xây dựng. Theo IUCN (2013),
Thông lá dẹt được đánh giá mức sắp bị tuyệt chủng (VU A2c B1ab (iii). Hiện nay,
các rừng Thông lá dẹt đang bị đe dọa nghiêm trọng do tình trạng phá rừng làm
nương rẫy, nhiều cây bị mất môi trường sinh sống tối ưu nên chết rụi, một số cây
quá già cũng tự đổ gãy. Tái sinh tự nhiên hầu như chỉ hạn chế ở giai đoạn cây mầm,
lại gặp chủ yếu ở nơi có khoảng trống, ven đường. Mặt khác lại thiếu vắng các cây
tái sinh ở tuổi trung gian, nên khó đủ sức thay thế những cánh rừng Thông lá dẹt cổ
thụ đang tồn tại [5], [6].
1.1.3. Tình hình phân bố Thông lá dẹt ở Tây Nguyên
Phân bố trong nước: Loài đặc hữu hẹp của tiểu vùng địa lý thực vật Nam
Trường Sơn, phân bố chủ yếu tại tỉnh Lâm Đồng và một số vùng giáp ranh thuộc
các tỉnh lân cận như Đắc Lắc, Khánh Hòa và Ninh Thuận.
7
Phân bố thế giới: không gặp bất kỳ nơi nào trên thế giới ngoài Việt Nam.
Thông lá dẹt thường mọc ở độ cao từ 1000 đến 2000 m so với mực nước
biển, trong những khu rừng khép tán thường xanh, xen với những cây họ Đậu và họ
Long não. Loài thường mọc trên đỉnh hoặc sườn núi nơi có lớp đất ẩm và tầng mùn
dày [4], [36]. Căn cứ vào các dẫn liệu thu thập được qua khảo sát, nghiên cứu ở các
điểm còn rừng nguyên sinh ở hầu khắp nước ta và đối chiếu với tiêu chuẩn của các
thứ hạng trong Danh lục đỏ của IUCN, thoạt đầu thấy vài tiêu chuẩn loài chưa đến
mức bị đe dọa tuyệt chủng (sự suy giảm quần thể trong quá khứ, hiện tại nhiều nhất
< 30%, EOO < 5.000 km2, AOO < 500 km2, số lượng tiểu quần thể ≤ 10, và hầu hết
các tiểu quần thể đều nằm trong các Khu bảo tồn thiên nhiên, Khu du lịch sinh thái
hay ở các vùng núi xa xôi chưa bị lâm tặc vào chặt trộm). Tuy nhiên trong khoảng 5
năm gần đây ở hai tiểu quần thể quan trọng nhất đều thuộc VQG Bi Đúp - Núi Bà
gồm các cây gỗ to lớn ở dọc đường giao thông mới xây dựng từ Khánh Hòa đi lên
Đà Lạt qua VQG Bi Đúp - Núi Bà và đường giao thông đang xây dựng ở Đông
Trường Sơn (từ Đà Lạt ra Quảng Nam) xuất hiện nguy cơ bị lâm tặc chặt hạ và vận
xuất nên ở thời điểm hiện tại việc xếp loài vào thứ hạng sẽ nguy cấp VU A2a,c,d,
A3a,c,d, B2a,c, C1 là hợp lý nhất [4].
1.2. Đa dạng di truyền quần thể thực vật
1.2.1. Khái niệm về quần thể thực vật
Quần thể được định nghĩa là tập hợp một nhóm cá thể của một loài trong một
nơi sống cụ thể, chúng độc lập với các quần thể khác về quan hệ sinh sản [1]. Di
truyền quần thể được truyền từ thế hệ này sang thế hệ khác, và bị ảnh hưởng bởi các
yếu tố như kích thước quần thể, sức sinh sản, khả năng sống sót, phương phức sinh
sản, trao đổi và đột biến di truyền. Kích thước quần thể là kết quả của sự tương tác
phức tạp liên quan đến các điều kiện môi trường sống và các đặc tính quần thể của
loài. Kích thước quần thể đóng vai trò quan trọng liên quan đến phương thức sinh
sản, di truyền và tiến hoá. Nguồn gốc tiến hoá thường liên quan đến cá thể lai (thế
hệ tiếp theo) trong quần thể và loài. Thụ phấn chéo có thể sản sinh những cá thể lai
đa dạng. Cấu trúc di truyền của những cá thể này đóng góp vào tính đa dạng trong
8
quần thể và duy trì khả năng thích nghi cao của quần thể trong nhiều môi trường
sống.
Tác động của con người đến môi trường sống thường dẫn đến sự phá vỡ cấu
trúc quần thể hình thành những quần thể nhỏ, cô lập và dẫn đến làm suy giảm khả
năng thích nghi của quần thể với môi trường sống. Trong quần thể, thế hệ sau được
sản sinh bằng thụ phấn cận noãn sẽ dẫn đến sự khác biệt về di truyền giữa các quần
thể là lớn và làm tăng tần số gen đồng hợp tử trong các quần thể nhỏ.
1.2.2. Tính đa dạng di truyền quần thể thực vật
Đa dạng sinh học thường đề cập đến mức độ khác nhau của các dạng sống bao
gồm các cá thể động vật, thực vật, vi sinh vật và được biểu hiện từ mức độ phân tử đến
hệ sinh thái. Công ước quốc tế về Đa dạng sinh học (1994) đã xác định đa dạng sinh
học bao gồm 3 cấp: (i) đa dạng trong loài (đa dạng di truyền hay đa dạng nguồn gen),
(ii) đa dạng giữa các loài (đa dạng về thành phần loài hay đa dạng loài) và (iii) đa dạng
hệ sinh thái. Đa dạng sinh học là kết quả của quá trình tiến hoá tự nhiên trải qua hàng
triệu năm, bao gồm cả 3 mức độ hệ sinh thái, loài và di truyền. Lý thuyết tiến hoá trên
cơ sở chọn lọc tự nhiên của Darwin đã dự đoán rằng đa dạng di truyền là thành phần
chính của đa dạng sinh học. Đa dạng di truyền hay còn gọi là đa dạng gen (đa dạng
DNA), là tập hợp những biến đổi của các gen và các kiểu gen trong nội bộ của một
loài. Đây là sự đa dạng quan trọng nhất, quyết định một loài có thể tồn tại lâu dài trong
tự nhiên hay không. Tính đa dạng này, vì thế đã và đang là nguồn cung cấp vật liệu
cho các chương trình chọn tạo và cải tiến giống [1]. Đa dạng di truyền cho phép các cá
thể và loài xử lý trước những biến đổi bất lợi của môi trường sống và có khả năng tự
phục hồi trong môi trường sống của chúng. Như vậy, đa dạng di truyền được đề cập
đến như là mức độ đa hình của mỗi cá thể trong suốt thời gian sống của nó, hoặc được
phản ánh bởi số alen của quần thể tại một vị trí địa lý trong một khoảng thời gian cụ
thể hoặc số alen của một loài trong phạm vi phân bố địa lý và lịch sử tồn tại của nó.
1.2.3. Một số kỹ thuật sinh học phân tử trong nghiên cứu đa dạng di truyền ở thực
vật
a) Kỹ thuật isozyme
9
Kỹ thuật isozyme là kỹ thuật nghiên cứu sự đa hình enzyme. Phương pháp
này được Hunter và Market đưa ra từ năm 1957, sau đó Harris hoàn thiện vào năm
1966 và bắt đầu được sử dụng phổ biến từ thập niên 70 đến nay. Mặc dù hiện nay
có nhiều kỹ thuật phân tử phát triển nhưng kỹ thuật isozyme vẫn được sử dụng vì
cách thức thực hiện tương đối nhanh, chi phí thấp, thích hợp cho các nghiên cứu xác
định mức độ biến đổi di truyền ở cấp độ thấp. Ngoài ra việc kết hợp kỹ thuật
isozyme với các kỹ thuật nghiên cứu đa hình DNA cho phép phân tích, so sánh
những đặc tính bền vững (hoặc thay đổi) theo điều kiện khác nhau của môi trường
[9].
b) Kỹ thuật RAPD (Random Amplified Polymorphic DNA)
Kỹ thuật RAPD được hai nhóm nghiên cứu của Welsh và Clelland (1991) và
Williams và cộng sự (1990) đồng thời xây dựng [70], [71]. Kỹ thuật này dựa trên
ứng dụng kỹ thuật PCR sử dụng các mồi đơn có trình tự ngẫu nhiên gồm khoảng 10
nucleotide. Số lượng các đoạn DNA được nhân bản phụ thuộc vào độ dài và vị trí
các đoạn mồi, kích thước và cấu trúc DNA genome. Sự đa hình của kỹ thuật RAPD
nói chung là do đột biến ở vị trí gắn mồi làm ngăn trở đến việc bắt cặp của mồi. Kết
quả là sau khi điện di sản phẩm RAPD – PCR sẽ phát hiện được sự khác nhau trong
phổ các phân đoạn DNA được nhân bản. Sự khác nhau đó gọi là tính đa hình chiều
dài sản phẩm PCR.
Kỹ thuật RAPD thao tác nhanh, đơn giản, giá thành thấp, dễ thực hiện do
không cần biết trước trình tự bộ gen của đối tượng cần nghiên cứu, sử dụng lượng
nhỏ DNA khuôn, chất lượng DNA khuôn không cần độ tinh sạch quá cao. Tuy
nhiên chỉ thị RAPD là chỉ thị trội do đó những kiểu gen lặn quy định tính trạng nào
đó sẽ khó phát hiện sự đa hình khi điện di sản phẩm [13], [72]. Kỹ thuật RAPD có
độ chính xác không ổn định (thể hiện ở mức độ lặp lại giống nhau thấp) [9], [10].
c) Kỹ thuật RFLP (Restriction Fragment Length Polymorphism)
Đây là kỹ thuật dựa trên tính đa hình chiều dài các đoạn cắt giới hạn, do
Botstein và cộng sự (1980) xây dựng vào năm 1980 để lập bản đồ di truyền ở người
[15]. Kỹ thuật này dựa trên đặc điểm của các enzyme giới hạn khác nhau, tạo nên
10
các đoạn cắt DNA khác nhau phân biệt được bằng điện di đồ. Số lượng các đoạn
này phụ thuộc vào số điểm nhận biết của enzyme giới hạn trong hệ gen, các đoạn
cắt còn được gọi là các “dấu vân tay” đặc trưng cho từng phân tử DNA. DNA của
mẫu nghiên cứu sau khi được tách chiết và tinh sạch sẽ được cắt với cùng một số
loại enzyme giới hạn. Tính đa hình độ dài được phát hiện nhờ đánh dấu phóng xạ
các mẫu dò DNA bổ sung tạo ra từ cùng một locus. Chỉ thị RFLP thường được ứng
dụng trong một số lĩnh vực như: nghiên cứu sự đa dạng của bộ genome, xây dựng
bản đồ di truyền, xác định mức độ liên kết với các tính trạng nông sinh học, xác
định con lai F1 trong lai hữu tính và lai tế bào trần, cũng như khả năng có chứa gen
bất dục tế bào chất, bất dục đực nhân mẫn cảm với nhiệt độ và ánh sáng, tìm dãy
alen tự bất hợp, gen phục hồi và gen duy trì. Tuy nhiên kỹ thuật này có nhược điểm
là quy trình phức tạp, cồng kềnh, tốn kém và sử dụng chất đồng vị phóng xạ gây
nguy hiểm cho người thao tác thí nghiệm.
d) Kỹ thuật AFLP (Amplified Fragment Length Polymorphism)
Kỹ thuật AFLP được hiểu là sự đa dạng của các đoạn DNA được nhân lên có
định hướng sau khi bị cắt bởi các enzyme giới hạn. Nguyên tắc của kỹ thuật này
tương tự như kỹ thuật RAPD nhưng mồi được sử dụng ở đây bao gồm một phần cố
định dài hơn (5bp) chứa vị trí nhận biết của enzyme giới hạn và một phần thay đổi
ngắn (2-4bp). Phần cố định dài tạo ra sự ổn định của sản phẩm được nhân lên, phần
thay đổi ngắn sẽ tạo ra nhiều locus, có thể lên đến hơn 100 locus được nhân lên với
mồi AFLP đơn. AFLP phát hiện sự khác nhau của các đoạn DNA bởi sự nhân có
chọn lọc các trình tự DNA hệ gene đã được cắt (bởi enzyme giới hạn) và được gắn
với đoạn tiếp hợp (adapter). Phương pháp cho phép sàng lọc đồng thời một số lượng
lớn các chỉ thị không có nghĩa. Sự đa hình được xác định bởi sự có mặt hoặc không
có mặt của một phân đoạn DNA. Phương pháp AFLP kết hợp được những ưu điểm
của RFLP và RAPD nên có hiệu quả trong phát hiện và phân tích đa hình một cách
nhanh chóng, ổn định và đáng tin cậy. Tuy nhiên chi phí và yêu cầu chuyên môn kỹ
thuật để thực hiện phân tích AFLP là rất cao nên phương pháp này ít được sử dụng
để nghiên cứu phân loại học phân tử mà chủ yếu được sử dụng trong việc thiết lập
bản đồ di truyền các tính trạng số lượng [72].
11
e) Kỹ thuật SSR (Simple Sequence Repeat)
Kỹ thuật SSR hay microsaterlite (vi vệ tinh) là kỹ thuật nghiên cứu dựa trên
trình tự lặp các đoạn đơn giản trên genome của sinh vật, đây là những trình tự ngắn
(từ 2 đến 6 cặp bazơ, ví dụ (AT)n; (AG)n; (AGTC)n) có thứ tự lặp lại liên tiếp dao
động từ 2 đến 40 đơn vị. Các trình tự lặp đơn giản rất phổ biến ở hệ gen động vật và
thực vật, mật độ các trình tự dao động rất lớn, phân bố trong hệ gen và có tính đặc
trưng cho từng loài [9].
Kỹ thuật này dựa trên nguyên lý phản ứng chuỗi PCR với mục tiêu đầu tiên
là nhận dạng các trình tự lặp lại đơn giản. Sau khi các trình tự lặp lại đơn giản này
được nhận dạng, bước tiếp theo là xác định trình tự của DNA ở hai đầu của các
đoạn lặp lại và thiết kế mồi. Các trình tự gần kề và các trình tự lặp lại sẽ tạo nên
SSR. Mồi SSR sau đó được sử dụng tương tự như các mồi RAPD. Kỹ thuật SSR có
tiềm năng rất lớn do có khả năng phát hiện tính đa hình rất cao, có thể phân biệt
được sự sai khác mà không xác định được bằng các chỉ thị khác như RAPD và
AFLP. Phản ứng không quá tốn kém, tiết kiệm được thời gian và hoá chất. Vì vậy
SSR là công cụ hữu hiệu trong nghiên cứu đa dạng di truyền nguồn gen cũng như
lập bản đồ hệ gen ở sinh vật [53]. Mồi sử dụng trong kỹ thuật SSR dài hơn mồi
RAPD và dựa trên trình tự đặc trưng của loài, vì thế độ tin cậy cao hơn so với kỹ
thuật RAPD. Hơn nữa mồi SSR là các locus đặc trưng, nên cung cấp nhiều thông tin
rất có ích cho việc phát hiện sự thay đổi các trình tự hiếm. SSR là loại chỉ thị đồng
trội nên đã nhanh chóng thay thế chỉ thị AFLP và RAPD và trở thành công cụ hữu
hiệu trong các ứng dụng chọn giống thực vật và nghiên cứu di truyền.
Nhược điểm của kỹ thuật này là chi phí cao và khó khăn trong việc thiết kế
cặp mồi đặc hiệu cho một locus đa hình. Để xây dựng cặp mồi đặc hiệu cần tách
dòng và đọc trình tự một số lượng lớn các đoạn DNA hệ gen chứa đoạn lặp lại. Một
vấn đề khác cũng thường gặp trong sử dụng SSR là việc xác định quan hệ giữa các
alen với các chỉ thị phân tử là rất khó. SSR có thể được phân bố ngẫu nhiên trong
genome nhưng cũng có khi tập trung lại ở tâm động hay eo thứ cấp của nhiễm sắc
thể. Điều này hạn chế việc sử dụng các mẫu dò nhiều locus trong phân tích liên kết
di truyền và nghiên cứu quần thể [9].
12
f) Kỹ thuật ISSR (Inter Simple Sequence Repeat)
Kỹ thuật do Zietkiewicz và cộng sự phát hiện năm 1994 [76]. Trong cấu trúc
hệ gen của vi sinh vật nhân thật tồn tại một loại các trình tự nucleotide lặp lại,
chúng thường đặc trưng cho loài. Trình tự này gồm từ 2 đến 5 nucleotide lặp lại
nhiều lần, ví dụ: (AT)n, (AG)n, (AGTC)n và nằm rải rác trong hệ genome của thực
vật bậc cao. Kỹ thuật ISSR dựa trên kỹ thuật PCR và dùng một mồi đơn có độ dài
từ 20 đến 30 nucleotide để nhân bản các trình tự đơn giản ở giữa các trình tự lặp lại
cho loài. Đoạn mồi được thiết kế là đoạn oligonucletide có trình tự bổ sung với trình
tự lặp. Khoảng cách giữa các trình tự lặp khác nhau làm cho các phân đoạn DNA
được nhân có độ dài ngắn khác nhau và đặc trưng cho mỗi cá thể. Nhiều kết quả
nghiên cứu đã chỉ ra chỉ thị ISSR phản ánh mức độ phân nhóm di truyền của quy
luật Mendel [66]. Chỉ thị này cũng được sử dụng rất hiệu quả trong nghiên cứu đa
dạng DNA ở một số đối tượng cây trồng [24], [35], [43], [48].
1.3. Ý nghĩa của nghiên cứu cứu xác định trình tự nucleotide ở thực vật
1.3.1. Ý nghĩa của nghiên cứu giải mã trình tự nucleotide ở thực vật
Nghiên cứu giải mã một số vùng gen bảo thủ cho mục đích phân loại, nhận
dạng một số nguồn gen quý là hướng nghiên cứu được phát triển mạnh trên thế giới
trong những năm gần đây. Cơ sở của phương pháp này là dựa trên kỹ thuật phân
tích DNA còn được gọi là “dấu vân DNA- DNA fingerprint”. Ứng dụng hướng
nghiên cứu này giúp nhận biết ranh giới các loài (giám định gen); đánh giá sự phát
sinh chủng loại giữa các loài (tiến hóa loài) [28]. Phương pháp được xây dựng dựa
trên thành phần và cấu trúc của các vùng gen đặc hữu của các taxon sinh vật, tập
trung vào hai vùng gen chính là gen nhân (ITS) và vùng gen lục lạp (cpDNA, nên có
hiệu quả cao trong việc định loại và giám định loài. Với các giá trị khoa học nêu
trên, đến nay việc xác định trình tự nucleotide vùng gen đặc trưng đang là công cụ
hỗ trợ đắc lực cho các nhà nghiên cứu trong việc phát hiện các loài mới, giải quyết
các mối nghi ngờ về vị trí phân loại, đánh giá đầy đủ về tính đa dạng di truyền, quan
hệ chủng loại và mức độ tiến hoá của nhiều loài động thực vật và vi sinh vật. Hiện
nay, ở Việt Nam hướng nghiên cứu này đang được sử dụng trong nghiên cứu phân
13
loại, định loại, tiến hóa,… trên nhiều đối tượng sinh vật ở Viện Công nghệ sinh học,
Viện Sinh thái và Tài nguyên sinh vật, Bảo tàng Thiên nhiên Việt Nam,…
1.3.2. Giới thiệu tổng quát về ba vùng gen giải mã trình tự nucleotide
a) Vùng gen thuộc hệ gen lục lạp
Hệ gen lục lạp (cpDNA) là một phân tử DNA vòng, sợi đơn, mỗi gen thường
không lặp lại, có kích thước từ 120 kb - 220 kb. Không giống như các gen nhân, các
gen lục lạp chỉ mã hoá các protein cần thiết cho chức năng quang hợp. Hệ gen lục
lạp thường được sử dụng cho nghiên cứu phân loại ở thực vật do đặc tính di truyền
theo dòng mẹ, không bị tái tổ hợp di truyền cho thế hệ sau và rất bảo thủ [55]. Hệ
gen lục lạp được đánh giá là sự tích lũy các đột biến theo thời gian, nên phản ánh
đúng mức độ tiến hóa của loài. Các gen lục lạp có tốc độ đột biến thấp hơn từ 4 - 5
lần so với gen trong nhân, nhưng nhanh hơn khoảng ba lần so với DNA ty thể thực
vật và thường xuyên được sử dụng trong nghiên cứu phân loại. Hiện nay, các vùng
gen matK, trnL - trnF, vùng đệm psbA - trnH, rpoC2…hay được sử dụng trong
nghiên cứu hệ thống học phân tử thực vật. Tất cả các gen thuộc hệ gen lục lạp
thường có mức độ biến đổi không lớn hơn 2 % giữa các loài lân cận.
- Vùng psbA - trnH: thường được sử dụng cho nghiên cứu phân loại [58].
Vùng này có kích thước xấp xỉ 450 bp, xác suất nhân bản thành công rất cao (100 %
với các loài đã được nghiên cứu). Mức độ khác biệt trình tự nucleotide giữa các loài
trung bình là 1,24 % và sự khác biệt bên trong loài rất thấp từ 0 – 0,08 % [31].
Trình tự psbA - trnH cũng đã được công bố trên ngân hàng gen với nhiều loài khác
nhau thuộc thực vật hạt trần, dương xỉ, rêu và rêu tản.
- Gen trnL: Là gen mã hoá cho tRNA vận chuyển Leucine trong lục lạp, có
kích thước khoảng 440-750 bp. Gen này được sử dụng nhiều trong nghiên cứu phân
loại phân tử [30], [42], [59]. Các kết quả thu được trong các nghiên cứu nguồn gốc
phát sinh loài sử dụng gen trnL cho thấy đây là một vùng DNA hữu ích cho phân
loại.
b) Vùng gen nhân (ITS)
Vùng ITS (Internal Transcribed Spacer) của gen mã hoá cho ribosome nhân
gồm các đơn vị gen 18S, 5,8S và 26S. Giữa các đơn vị gen có các đoạn ITS-1 và
14
ITS-2, các thành phần này tạo thành một nhóm gen cơ bản. Các nhóm gen như vậy
lặp lại liên tục trong hàng nghìn bản sao trong hệ gen nhân và chúng được ngăn
cách bởi vùng NTS (nontranscribed spacer).
Trong các nghiên cứu phân loại thực vật ở mức độ loài, vùng ITS là locus
được xác định trình tự phổ biến nhất, được kiến nghị làm vùng DNA barcode cho
thực vật [12], [14]. Ở mức độ loài, vùng ITS có mức độ đa dạng cao (khoảng 13,6 %
giữa các loài gần gũi), nhưng lại có mức độ biến đổi thấp bên trong loài [14]. Với sự
hiện diện của trên 70.000 trình tự ITS được công bố trên ngân hàng Genbank năm
2011 đây là nguồn tư liệu có giá trị, mở ra những triển vọng lớn cho nghiên cứu
phân loại và giám định.
1.4. Một số thành tựu nghiên cứu tính đa dạng di truyền và tiến hóa phân tử ở
các loài lá kim
1.4.1. Trên thế giới
Do có giá trị kinh tế và dược liệu cao, nên các loài lá kim nói chung trên thế
giới đã bị khai thác và tàn phá nhiều. Vì thế tính đa dạng của chúng đã và đang bị
đe dọa cả ở mức độ loài và quần thể. Việc nghiên cứu di truyền quần thể phục vụ
cho công tác bảo tồn và phục hồi các loài Thông trên thế giới đang là “điểm nóng”
và được nhiều nhà nghiên cứu trên thế giới quan tâm [25].
Năm 2010, Wang và Hao đã sử dụng chỉ thị ISSR để đánh giá đa dạng di
truyền của 13 quần thể thông tự nhiên ở Trung Quốc (Pinus tabulaeformis Carr.)
[69]. Kết quả chỉ ra rằng tổng số biến đổi di truyền trong quần thể Thông ở Trung
Quốc chủ yếu duy trì bên trong quần thể. Sự đa dạng di tuyền có xu hướng giảm từ
quần thể trung tâm đến quần thể trung gian và quần thể biên. Cả hai yếu tố phân bố
tự nhiên và hoạt động của con người đều ảnh hưởng đến sự hình thành cấu trúc di
truyền của loài này. Hay như nhóm tác giả Mariette và cộng sự (2001) bằng việc
phân tích hai chỉ thị AFLP và SSR đã đánh giá đa dạng di truyền của 23 quần thể
Pinus pinaster xuất xứ từ Aquitaine và Corsican thuộc nước Pháp [39]. Kết quả chỉ
ra rằng những quần thể Pinus pinaster xuất xứ từ Aquitaine có sự đa dạng di truyền
cao hơn so với những quần thể xuất xứ từ Corsican. Ngoài ra còn nhiều nhóm tác
khác trên thế giới cũng đã quan tâm và nghiên cứu vấn đề này như Kadermir và
15
cộng sự (2004) đã nghiên cứu đa dạng di truyền loài thông đỏ Thổ Nhĩ Kỳ [29];
Ledig và cộng sự (2005) nghiên cứu trên loài Picea breweriana [34]; Clark và cộng
sự (2000) đã nghiên cứu trên những loài thuộc chi Abies trong họ Thông [20],…
Đặc biệt thông qua việc phân tích cấu trúc di truyền sử dụng chỉ thị ISSR, AFLP,
của một số loài lá kim bị đe doạ tuyệt chủng cho thấy mức độ đa dạng di truyền bị
suy giảm rất cao liên quan đến khả năng tăng hệ số đồng hợp tử trong các quần thể
nhỏ và hẹp. Những nghiên cứu đó cũng chỉ ra mức độ đa dạng giữa các quần thể là
rất lớn và đồng thời đưa ra một số biện pháp hiệu quả để phục hồi nguồn gen một số
loài lá kim bị đe dọa tuyệt chủng [18], [73].
Riêng đối với loài Thông lá dẹt, công bố mới nhất về tính đa dạng nguồn gen
quần thể Thông lá dẹt của Việt Nam được xác định bởi nhóm nghiên cứu của Wang
và cộng sự (2014) [68]. Trong nghiên cứu này tác giả đã sử dụng 57 mẫu được lấy
từ sáu quần thể Thông lá dẹt tại Vườn Quốc gia (VQG) Bi Đúp để phân tích sự thay
đổi trình tự nucleotide tại mười vùng gen nhân (~ 9 kb), 14 vùng gen ti thể (~10 kb)
và bảy vùng gen lục lạp (cpSSR). Kết quả cho thấy mức độ đa dạng nucleotide rất
thấp tại các vùng gen nhân (0,0020). Trong khi đó, mức độ đa dạng gen dị hợp
(haplopype) của các vùng gen cpSSR tương đối cao (0,911), nhưng lại không có
tính đa hình nucleotide khi phân tích với tất cả 14 vùng gen ty thể (mtDNA). Trong
nghiên cứu này, các tác giả cũng đã chỉ ra mức độ sai khác di truyền quần thể ở 10
vùng gen nhân phân tích là rất thấp (5,2%) [68].
1.4.2. Trong nước
Việt Nam hiện được xếp vào một trong 10 điểm nóng nhất trên thế giới về
bảo tồn Thông nên việc nghiên cứu bảo tồn các loài lá kim cần được coi trọng [4],
[5]. Vì vậy, việc nghiên cứu tính đa dạng di truyền các loài lá kim có giá trị khoa
học, kinh tế, các loài đặc hữu, quý hiếm đang bị đe dọa tuyệt chủng ở Việt Nam là
hết sức cần thiết. Tuy nhiên ở Việt Nam hiện có rất ít công trình nghiên cứu đánh
giá mức đa dạng di truyền một số loài lá kim. Hiện nay ở Việt Nam chỉ có nhóm
nghiên cứu của Phòng Phân loại học thực nghiệm và Đa dạng nguồn gen thuộc Bảo
tàng Thiên nhiên Việt Nam (BTTNVN) là đơn vị đã và đang có những nghiên cứu
sâu về đa dạng di truyền nguồn gen một số loài Thông trên cơ sở phân tích phân tử.
16
Chẳng hạn như Vũ Đình Duy và cộng sự (2010) đã sử dụng chỉ thị ISSR và SSR để
đánh giá đa dạng di truyền của 4 loài thuộc họ Hoàng đàn (Cupressaceae) là Pơ mu
(Fokienia hodginsii), Sa mộc dầu (Cunninghamia lanceolata var. konishii), Hoàng
đàn hữu liên (Cupressus tonkinesis) và Thủy tùng (Glytostrobus pensilis) đã chỉ ra
hai loài Pơ mu và Sa mộc dầu có mức độ suy giảm cao hơn Thủy tùng và Hoàng
đàn hữu liên, sự suy giảm đều liên quan đến hoạt động của con người, đặc biệt nơi
sống bị phân cắt. Ngoài ra, bằng việc giải mã vùng gen 18S, nhóm tác giả đã giải
quyết vấn đề tồn tại về taxon của Sa mộc dầu (Cunninghamia lanceolata var.
konishii) [2]. Hay Nguyễn Minh Tâm và cộng sự (2010) đã giải mã vùng gen rpoC,
gen rbcL và gen matK để nghiên cứu mối quan hệ di truyền của 15 loài lá kim
thuộc lớp Thông (Thủy tùng, Thông đỏ bắc, Bách xanh núi đá, Bách xanh núi đất,
Hồng tùng, Thông Pà cò, Thông tre lá dài, Thông đà lạt, Thông nàng, Thông tre lá
ngắn, Thông ba lá, Kim giao nam, Kim giao bắc, Dẻ tùng vân nam và Thông đỏ
nam) và đã chỉ ra mối quan hệ gần gũi của các loài [8]. Để nghiên cứu mối quan hệ
họ hàng giữa các taxon trong thực vật, Tam và Trang (2012) đã sử dụng vùng gen
18S để xác định mối quan hệ tiến hoá của 6 chi thuộc họ Hoàng đàn (Cupressaceae)
ở Việt Nam [58]. Kết quả chỉ ra 2 nhánh tiến hoá có quan hệ mật thiết với nhau,
Xanthocyparis vietnamesis/Cupressus tonkinensis và Fokienia hodginsii/Cupressus
rupestris/C. formasana. Xanthocyparis noothatensis có quan hệ gần gũi với loài
thuộc chi Cupressus. Fokienia hodginsii cùng nhánh tiến hoá với chi Calocedrus.
Tương tự, Đinh Thị Phòng và cộng sự (2009), Vũ Thi Thu Hiền và cộng sự (2009)
cũng đã sử dụng chỉ thị thị RAPD, ISSR và cpSSR để nghiên cứu mối quan hệ di
truyền cho Pơ mu, Bách xanh phục vụ cho nghiên cứu bảo tồn [3], [7]. Đến nay có
thể nói, các dẫn liệu về đa dạng di truyền, các trình tự nucleotide đặc trưng cho một
số loài lá kim đặc hữu, có giá trị kinh tế cao và có nguy cơ tuyệt chủng hầu như
chưa được nghiên cứu hoặc rất hạn chế nên cần được quan tâm nghiên cứu để có kế
hoạch bảo tồn cũng như khai thác nguồn tài nguyên quý giá này.
17
CHƢƠNG II. VẬT LIỆU VÀ PHƢƠNG PHÁP NGHIÊN CỨU
2.1. Vật liệu nghiên cứu
Vật liệu nghiên cứu:
- Gồm 70 mẫu lá và gỗ của 70 cá thể từ 4 quần thể loài Thông lá dẹt (Pinus
krempfii) thu tại 2 tỉnh Lâm Đồng và Đắc Lắc. Các mẫu do TS. Nguyễn Tiến Hiệp,
cộng tác viên của Bảo tàng Thiên nhiên Việt Nam cung cấp. Mẫu lá, gỗ sau khi thu
được bảo quản trong sillicagel và giữ ở nhiệt độ phòng cho tới khi sử dụng. Thông
tin chi tết các mẫu nghiên cứu được thể hiện trong bảng 2.1.
- Bốn mẫu (Pk5; Pk8; Pk18 và Pk45) đại diện của 4 quần thể Thông lá dẹt để
xác định trình tự nucleotide 01 vùng gen nhân và 02 gen lục lạp.
Bảng 2.1. Thông tin của các mẫu thu nghiên cứu phân tử
Tên
quần
thể
Địa điểm
thu mẫu
Bi Đúp
Núi Bà,
Lâm Đồng
Bi Đúp
Cổng
Núi Bà,
Trời
Lâm Đồng
Bi Đúp
Hòn
Núi Bà,
Giao
Lâm Đồng
Chư Chư Yang
Yang Sin, Đắc
Sin
Lắc
Yang
Ly
Số
lƣợng
mẫu
Vị trí địa lí
Kí kiệu
mẫu
Vĩ độ bắc
(◦N)
Kinh độ đông
(◦E)
Độ cao
so mặt
nƣớc
biển (m)
5
Pk1- Pk5 12o11’02.7”N 108o41’24.3”E
1482 1485
11
Pk6-Pk16 14o26’34.0”N
108o34’3.0”E
952 1000
26
Pk17Pk42
12o11’03.2”N 108o41’30.3”E
14821593
28
Pk43Pk70
12o25’05.2”N 108o22’17.1”E
11101120
Các cặp mồi sử dụng trong nghiên cứu:
- Hai mươi sáu mồi ISSR và 17 cặp mồi SSR dùng để đánh giá đa dạng di
truyền quần thể Thông lá dẹt. Các cặp mồi được tổng hợp bởi hãng IDT (Intergrated
DNA Technology, Mỹ), có ký hiệu và trình tự nucleotide như trong bảng 2.2 và
bảng 2.3.
18
Bảng 2.2. Trình tự nucleotide của 26 mồi ISSR sử dụng trong nghiên cứu
Tên
mồi
Trình tự
nucleotide
1
HB12
(CAC)3GC
[37]
14
ISSR16
(CT)8AC
[45]
2
HB15
(GTG)3GC
[37]
15
ISSR18
(CT)8A
[27]
3
ISSR1
(CAG)5
[45]
16
ISSR52
(CT)8G
[69]
4
ISSR2
(CAA)5
[45]
17
ISSR55
(AC)8T
[69]
5
ISSR3
(GACA)4
[45]
18
ISSR61
(AC)8TG
[69]
6
ISSR5
(CCG)6
[27]
19
ISSR67
(ATG)6
[69]
7
ISSR6
(CTC)6
[27]
20
ISSR69
(GGGTG)3
[69]
8
ISSR7
(GGC)6
[27]
21
UBC811
(GA)8 C
[49]
9
ISSR8
(GAA)6
[69]
22
UBC834
(AG)8 CT
[49]
10
ISSR9
(TG)8GA
[69]
23
UBC843
(CT)8GA
[69]
11
ISSR10
(CTC)8
[69]
24
UBC851
(GT)8 CG
[69]
13
ISSR11
(CCA)5
[37]
25
UBC854
(TC)8AG
[69]
13
ISSR15
(CA)8A
[45]
26
UBC859
(TG)8 GC
[69]
STT
Tham
STT Tên mồi
khảo
Trình tự
nucleotide
Tham
khảo
Bảng 2.3. Trình tự nucleotide của 17 cặp mồi SSR sử dụng trong nghiên cứu
STT
Tên mồi
1
NZPRP6
2
Pt79951
3
Pt36480
4
P5
Trình tự nucleotide (5’ – 3’)
F GGAAGAAAAATTGGGCCTTA
R CTCTCTATCTCTGCCCCA
F CTTTTGTTTTTCAACAATTGCA
R ACATCTATCTCCCATATCGGC
F TTTTGGCTTACAAAATAAAAGAGG
R AAATTCCTAAAGAAGGAAGAGCA
F GTTCGCTAGTTTGTTTGATCCC
R TCCCAGCAAATCCTTGACTC
Tham khảo
[22]
[20]
[20]
[60]
19
5
Pt9383
6
PtTX3026
7
Pt26081
8
Pt30204
9
Pt71936
10
Pt87268
11
Pt15196
12
Pnh038
13
PeC26BGT
14
PRE24
15
PtTX3020
16
RPS2
17
RPS160
F AGAATAAACTGACGTAGATGCCA
R AATTTTCAATTCCTTTCTTTCTCC
F AATACTTGGGAGGGATAC
R AATAGCCAGTTTTGTTTG
F CCCGTATCCAGATATACTTCCA
R TGGTTTGATTCATTCGTTCAT
F TCATAGCGGAAGATCCTCTTT
R CGGATTGATCCTAACCATACC
F TTCATTGGAAATACACTAGCCC
R AAA ACC GTA CAT GAG ATT CCC
F GCCAGGGAAAATCGTAGG
R AGAAGATTAGACATCCAACCC
F CTTGGATGGAATAGCAGCC
R GGAAGGGCATTAAGGTCATTA
F ACTCATTTCCGGATGGTGG
R TGGGGCTTGGACTTCAAGA
F CATCCACCAGTTTCCCTACG
R ATTTGAACGCACAGAGCATTT
F GTTTTTTAAATTGGGAAGGCG
R CGTGGGGGAGATAGTGATAGAGT
F GTCGGGGAAGTGAAAGTA
R CTAGGTGCAAGAAAAGAGTAT
F CATGGTGTTGGTCATTGTTCCA
R TGGAGGCTATCACGTATGCACC
F ACTAAGAACTCTCCCTCTCACC
R TCATTGTTCCCCAAATCAT
[20]
[23]
[67]
[67]
[67]
[67]
[67]
[17]
[40]
[16]
[23]
[21]
[21]
- Ba cặp mồi đặc hiệu (01 cặp mồi nhân bản vùng gen nhân và 02 cặp mồi
nhân bản vùng gen lục lạp) sử dụng trong nghiên cứu như trong bảng 2.4. Trình tự
của các cặp mồi được tổng hợp bởi hãng IDT (Integrated DNA Technology, Mỹ).
20
Bảng 2.4. Kích thước lý thuyết của các cặp mồi dùng trong nghiên cứu
Vùng
gen
Ký hiệu
cặp mồi
Trình tự nucleotide (5’– 3’)
Kích
thƣớc
lý thuyết
(bp)
1100
Nhiệt độ
bắt cặp
(Tm)C
Nguồn
gốc tổng
hợp
ITS
ITSF/
ITSR
CCTGCGGAAGGATCATTGTC
TTAAACTCAGCGGGTAGTC
50
CGAAATCGGTAGACGCTACG
ATTTGAACTGGTGACACGAG
500
51
GTTATGCATGAACGTAATGCTC
CGCGCATGGTGGATTCACAATCC
600
51
Thiết kế
dựa vào
trình tự
nucleotide
loài
Elleanthus
conife mã
số
EU490666
Genbank
Taberlet et
al., (2006)
[61]
Sang et al.,
(1997) [50]
Tate et al.,
(2003) [64]
trnL trnF
trnLF/
trnFR
psbA
-trnH
psbA3'f/
trnHf
Các hóa chất dùng trong nghiên cứu:
Hóa chất tách chiết và tinh sạch DNA (CTAB, EDTA, Tris-HCl,
Isopropanol, ethanol, ARNase, chloroform, Kit tinh sạch genomic, Kit tinh sạch sản
phẩm PCR, đệm TAE, agarose, poly acrylamide, TE,...; các hóa chất dùng trong
phản ứng PCR: Đệm PCR, MgCl2, dNTP, Taq polymerase; hóa chất tinh sạch cản
phẩm PCR và xác định trình tự nucleotide: Quick Gel Extraction Kit QIAGEN, Dye
Teminator Cycle Sequencing Kit,… Các hóa chất sử dụng đều là của các hãng
Fermentas, Biobasis, QIAGEN, ...
:
), máy ly tâm
), bộ điện di Nytechnich (Anh), máy chụp ảnh gel (Cleaver, Đức)
máy ổn nhiệt
), giải mã trình tự trên máy ABI PRISM® 3100 Avant
Genetic Analyzer (Applied Biosystems),.…
Cối, chày sứ, thìa vô trùng, giấy thấm vô trùng, ống eppendorf, đầu côn các
loại,…của các hãng Canada, Mỹ, Trung Quốc và Việt Nam.
21
2.2. Nội dung nghiên cứu
- Xác định và phân tích các thông số di truyền của 4 quần thể Thông lá dẹt sử
dụng chỉ thị ISSR và SSR
- Xác định, phân tích và so sánh trình tự nucleotide của 03 vùng gen (01
vùng gen nhân và 02 vùng gen lục lạp) với các trình tự liên quan trên ngân hàng
Genbank
2.3. Địa điểm nghiên cứu
Nghiên cứu được tiến hành tại Phòng Phân loại học thực nghiệm và Đa dạng
nguồn gen - Bảo tàng Thiên nhiên Việt Nam - 18 Hoàng Quốc Việt, Cầu Giấy, Hà Nội.
2.4. Phƣơng pháp nghiên cứu
2.4.1. Tách chiết DNA tổng số từ 70 mẫu Thông lá dẹt
DNA tổng số được tách chiết từ lá hoặc vỏ cây bằng phương pháp của
Porebski và cộng sự (1997) [51]. Kiểm tra độ sạch trên gel agarose 0,9 % và đo
nồng độ DNA tổng số trên máy UVS 2700, Labomed, Hoa Kỳ.
2.4.2. Thiết kế cặp mồi
Để thiết kế được cặp mồi có tính đặc hiệu cao và giảm thiểu khả năng bắt cặp
không đặc hiệu. Chúng tôi đã khai thác các dữ liệu trong ngân hàng gen Genbank để
tìm ra tất cả các trình tự gen mã hoá cho vùng gen ITS đối với loài lá kim. Sau đó sử
dụng phần mềm BioEdit để so sánh, vùng bảo thủ nhất được sử dụng để thiết kế cặp
mồi. Cặp mồi ITSF/ITSR nhân bản vùng gen ITS được thiết kế dựa vào trình tự
nucleotide loài Elleanthus conifer mã số EU490666 trên ngân hàng Genbank. Quá
trình thiết kế cặp mồi được thực hiện trên phần mềm DNAstar. Hai cặp mồi
psbA3'f/trnHf và trnLF/trnFR được tổng hợp dựa trên những nghiên cứu của Sang
và cộng sự (1997), Tate và cộng sự (2003) và Taberlet và cộng sự (2006).
2.4.3. Nhân bản DNA
Phản ứng PCR (polymerase chain reaction) với các chỉ thị ISSR và SSR:
Phản ứng nhân gen được thực hiện trên máy PCR system 9700 (Hoa Kỳ) với tổng
thể tích 25 µl gồm các thành phần: dung dịch đệm PCR 1X; MgCl2 2,5 mM; dNTPs
22
2 mM; mồi 100 nM; 50 ng DNA khuôn và 0,5 đơn vị Taq polymerase. Chu trình
nhiệt của phản ứng PCR: biến tính ở 94°C trong 4 phút, tiếp sau là 35 chu kỳ nối
tiếp nhau với các bước: biến tính 94°C trong 1 phút; gắn mồi 50 - 58°C trong 1 phút
(đối với chỉ thị SSR), 46 – 52oC trong 1 phút (đối với chỉ thị ISSR); tổng hợp sản
phẩm 72oC trong 1 phút và kết thúc phản ứng ở 72°C trong 10 phút; giữ sản phẩm ở
4°C. Sản phẩm PCR được kiểm tra trên gel polyacrylamide 5 % cùng với thang
DNA chuẩn 100 bp (của hãng Fermentas), và gel agarose 1,5 % với thang chuẩn
1kb (của hãng Fermentas) sau đó nhuộm ethidium bromide 15 phút và quan sát dưới
tia UV.
Nhân bản DNA và đọc trình tự: phản ứng PCR được tiến hành với thể tích
25µl, gồm các thành phần: dung dịch đệm PCR 1X, MgCl2 2,5 mM, dNTPs 2 mM;
0,5 pMl cho một mồi xuôi và ngược, 0,5 đơn vị Taq polymerase và 50 ng DNA
tổng số. Nhân bản DNA được tiến hành trên máy PCR systems 9700 theo chu trình
sau: (1) 94oC: 3 phút; (2) 94oC: 1 phút, 50oC - 51oC: 1 phút, (4) 72oC: 1 phút, lặp lại
35 chu kỳ từ (2) đến (4) và (5) 72oC: 10 phút. Điện di sản phẩm PCR trên gel
agarose 1,5%.
2.4.4. Thôi gel và tinh sạch sản phẩm PCR
Tinh sạch sản phẩm PCR là bước quan trọng để thực hiện phản ứng giải trình
tự. Quá trình tinh sạch nhằm thu được sản phẩm đoạn gen mong muốn. Sản phẩm
PCR được điện di trên gel agarose 1,5 %, cắt lấy phân đoạn DNA quan tâm trên bàn
soi UV và tinh sạch bằng bộ KIT QIAquick Gel Extraction của QIAGEN. Kiểm tra
sản phẩm thu được trên gel agarose 0,9 %. Chụp ảnh sản phẩm thôi gel và xác định
hàm lượng DNA để gửi đi xác định trình tự.
2.4.5. Giải trình tự nucleotide các đoạn DNA
Các sản phẩm PCR sau khi tinh sạch được gửi sang công ty Macrogen (Hàn
Quốc) để xác định trình tự nucleotide theo hai chiều (xuôi và ngược).
2.5. Phân tích số liệu
- Phân tích đa dạng di truyền
Các thông số đa dạng di truyền ở mức độ quần thể và loài của loài Thông lá
dẹt: số alen trung bình (Na), số alen hiệu quả (Ne), và các thông số đa dạng di
23
truyền quần thể như phần trăm lô cút đa hình (PPB), chỉ số đa dạng di truyền
Shannon (I), hệ số gen dị hợp tử mong đợi (He), được xác định cho cả chỉ thị ISSR
và SSR bằng phần mềm GenAlex 6.3 [50].
Hàm lượng thông tin đa hình (PIC) của mỗi chỉ thị ISSR được xác định theo
công thức: PICi = 1 - ∑Pij2. Trong đó Pij là tần số alen thừ j của kiểu gen i được
kiểm tra. Phạm vi giá trị PIC từ 0 (không đa hình) tới 1 (đa hình hoàn toàn). Chỉ số
đa dạng di truyền (h) theo Nei (1973) của mỗi quần thể Thông lá dẹt với chỉ thị
ISSR được tính theo công thức: h = ∑pi2 (trong đó pi là tần số của alen thứ i tại
locus đó) [44].
Hệ số gen dị hợp tử quan sát (Ho), số alen hiếm (Ap) của mỗi quần thể; hệ số
khác biệt di truyền (Fst), mức độ di nhập gen (Nm) của mỗi locus SSR và phân tích
mức biến lượng phân tử (AMOVA) của quần thể được tính toán dùng phần mềm
GENALEX 6.3 [50]. Ma trận khoảng cách di truyền được thiết lập để phân tích
thành phần tọa độ (PCA) giữa các quần thể. Lập biểu đồ hình cây theo phương pháp
của Nei (1973) trong phần mềm NTSYS 2.0, version 2.0 [52], giá trị Bootstrap
được hỗ trợ bởi phần mềm Win-Boot [75] với số lần lặp lại 1000 lần
- Xác định trình tự nucleotide
Dữ liệu được xử lý bằng các phần mềm chuyên dụng BioEdit, Mega 4.0,
Clustal X 2.1, DNAstar,…vv. Trình tự nucleotide của loài Thông lá dẹt được so
sánh với các trình tự của vùng gen ITS, trnL-trnF và psbA-trnH đã công bố trên
Genbank.
Xây dựng cây phát sinh chủng loại: Có 4 phương pháp tạo cây bao gồm: (1)
phương pháp Neighbor – Joining (NJ), (2) phương pháp Minimum Evolution, (3)
phương pháp Maximum Parsimony và (4) phương pháp UPGMA, trong đó
Neighbor - Joining là phương pháp thường được sử dụng nhất. Khoảng cách di
truyền giữa các loài cũng được ước lượng thông qua độ dài của cành cây. được xây
dựng theo phương pháp NJ (Neighbor Joining) [58].
Dữ liệu DNA sau khi thực hiện ghép nối, xắp xếp thẳng hàng (alignment)
được tạo cây tiến hoá bằng phần mềm MEGA 4.0, sử dụng phương pháp Neighbor
- Joining (NJ) với các thông số phân tích như sau:
24
- Kiểm tra phân tích: kiểm tra bằng giá trị bootstrap với số lần lặp lại
(replicate) là 1000 lần.
- Dữ liệu thiếu/ khoảng trống nucleotide (do đột biến thêm bớt tạo ra): được
xoá bỏ theo cặp (pairwise deletion).
- Mô hình đột biến: mô hình Maximum composite Likelihood với số lượng đột
biến được tính bao gồm cả đột biến hoán đổi (A↔T, G↔C) và hoán vị (A↔C, A
↔ G, T ↔ C, T↔ G).
25
CHƢƠNG III. KẾT QUẢ NGHIÊN CỨU
3.1. Kết quả tách chiết DNA tổng số loài Thông lá dẹt
DNA tổng số của 70 cá thể từ 4 quần thể Thông lá dẹt (Pinus krempfii) được
tách chiết theo phương pháp của Porebski và cộng sự (1997) có cải tiến một số bước
cho phù hợp với phòng thí nghiệm [51]. Kết quả kiểm tra DNA tổng số trên gel
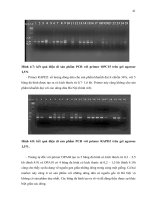
agarose 0,9 % (hình 3.1) cho thấy mỗi giếng chỉ cho một băng vạch duy nhất, các
băng đậm, sắc nét, thể hiện DNA tách chiết được có độ tinh sạch cao, không bị đứt
gãy. Kết quả này một lần nữa khẳng định chất lượng DNA tách chiết được hoàn
toàn đủ điều kiện cho những nghiên cứu tiếp theo.
1 2 3 4 5 6
7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36
Hình 3.1. Hình ảnh DNA tổng số đại diện của một số mẫu Thông lá dẹt (các giếng
từ 1 đến 36 là thứ tự các mẫu Thông lá dẹt như bảng 2.1).
DNA tổng số sau khi tách chiết được kiểm tra hàm lượng và độ sạch bằng
phương pháp đo quang phổ hấp thụ ở bước sóng 260 nm và 280 nm. Kết quả thu
được cho thấy, nồng độ DNA của 70 mẫu Thông lá dẹt dao động từ 640 đến 1000
ng/µl. Tỷ lệ OD260/OD280 thể hiện độ tinh sạch của các mẫu DNA dao động từ 1,732
đến 2 (số liệu không chỉ ra ở đây). Kết quả trên khẳng định các mẫu DNA tách chiết
được hoàn toàn đủ tiêu chuẩn cho những phân tích tiếp theo.
3.2. Tính đa dạng di truyền quần thể loài Thông lá dẹt
Trong nghiên cứu này, chúng tôi đã sử dụng 26 mồi ISSR và 17 cặp mồi
SSR để đánh giá tính đa dạng di truyền của bốn quần thể Thông lá dẹt thu ở Yang
Ly, Cổng Trời, Hòn Giao của tỉnh Lâm Đồng và Chư Yang Sin của tỉnh Đắc Lắc.
3.2.1. Tính đa dạng di truyền quần thể Thông lá dẹt phân tích với chỉ thị ISSR
Tính đa hình DNA giữa 70 mẫu Thông lá dẹt
Hai mươi sáu mồi ISSR đã được sử dụng để phân tích cho 70 cá thể thuộc
bốn quần thể Thông lá dẹt thu tại Yang Ly, Cổng Trời, Hòn Giao và Chư Yang Sin