Luận văn : BƯỚC ĐẦU XÂY DỰNG QUY TRÌNH ĐỊNH LƯỢNG CÁC SẢN PHẨM BIẾN ĐỔI GEN BẰNG PHƯƠNG PHÁP REAL-TIME PCR part 8 ppsx
Bạn đang xem bản rút gọn của tài liệu. Xem và tải ngay bản đầy đủ của tài liệu tại đây (398.15 KB, 13 trang )
78
78
Trong thí nghiệm này, chúng tôi sử dụng đối tượng là các mẫu được sàng lọc dương tính với
promoter 35S ở thí nghiệm 2. Các mẫu này được lặp lại 2 lần trong cùng một lần thí nghiệm. Các kết
quả được phân tích trên đồ thị DNA quantification như sau.
Hình 4.13: Đồ thị định lƣợng p35S của các mẫu thí nghiệm
Ghi chú:
Hình 4.14: Đƣờng chuẩn phản ứng định lƣợng p35S
Ghi chú:
B1 B2 B4 B6
B7 B10 B11
Mẫu chuẩn
Mẫu chưa biết
79
79
Hình 4.15: Đồ thị định lƣợng gen tham chiếu của các mẫu thí nghiệm
Ghi chú:
Hình 4.16: Đƣờng chuẩn phản ứng định lƣợng gen tham chiếu
Ghi chú:
Bảng 4.14: Các thông số kết quả của thí nghiệm
Thí nghiệm định lƣợng
Thông số
p35S
Gen tham
chiếu
Hệ số tƣơng quan (Correlation
Coefficient)
Hiệu quả PCR (PCR Efficiency) (%)
0,998
110,7
0,987
116,8
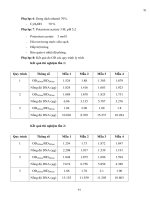
Bảng 4.15: Chu kỳ phát hiện của các mẫu thí nghiệm
B1 B2 B4 B6
B7 B10 B11
Mẫu chuẩn
Mẫu chưa biết
80
80
STT
Tên và đặc điểm của mẫu
Kí
hiệu
Chu kỳ phát hiện
Promoter
35S
Gen tham
chiếu
1
2
3
4
5
6
7
Bắp giống 1
Bắp giống 2
Bắp giống 4
Bắp trái ăn tươi
Bột bắp nguyên liệu làm thức
ăn gia súc
Thức ăn gia súc 2
Bột ngũ cốc dinh dưỡng 1
B1
B2
B4
B6
B7
B10
B11
27.38
27.61
28.93
29.73
26.35
26.51
34.16
33.46
27.71
27.8
26.05
27.33
24.99
25.81
27.69
27.14
25.6
24.4
23.02
24.23
23.91
23.93
28.32
28.31
24.92
24.92
30.59
27.44
Từ các kết quả trên đây chúng tôi rút ra một số nhận xét sau :
- Nhìn trên đồ thị DNA Quantification ta thấy các mẫu đều dương tính rõ rệt, các đường cong
đồ thị đều vượt lên trên khá rõ so với chu kỳ ngưỡng.
- Hiệu quả của phản ứng : Phản ứng đạt hiệu quả cao với đường chuẩn được xây dựng chính
xác thể hiện qua các thông số như : hệ số tương quan của đường chuẩn đạt 0,998 (định
lượng p35S) và 0,987 (định lượng gen tham chiếu) cao hơn yêu cầu của nhà sản xuất (0,98);
hiệu quả PCR cũng đạt 110,7 % (định lượng p35S) và 116,8 % (định lượng gen tham
chiếu).
Bảng 4.16: Độ lệch tiêu chuẩn chu kỳ phát hiện của mẫu
(C
T
Standard Deviation)
Mẫu
Độ lệch tiêu chuẩn của
chu kỳ phát hiện
(C
T
Standard Deviation)
p35S
Gen tham chiếu
81
81
B1
B2
B4
B6
B7
B10
B11
0.16
0.56
0.11
0.50
0.06
0.91
0.58
0.39
0.85
0.86
0.01
0.01
0.00
2.23
- Chu kỳ phát hiện của các mẫu thí nghiệm trong 2 lần lặp lại là xấp xỉ nhau. Điều này được
thể hiện ở Bảng 4.15 và 4.16 , độ lệch chuẩn của chu kỳ phát hiện là khá thấp, đặc biệt là
một số mẫu như B6, B7, B10.
- Tuy nhiên, nhìn trên đường chuẩn ở Hình 4.14 và 4.16, ta thấy một số mẫu có thể có kết
quả định lượng không chính xác do chúng vượt quá giá trị thấp nhất và cao nhất của các
mẫu chuẩn. Điều này có thể do sự ngoại nhiễm trong thao tác hay chất lượng DNA ly trích
gây ra.
Kết quả định lượng và tỷ lệ phần trăm chuyển gen trong các mẫu theo công thức bên dưới:
%GMO = (Số lƣợng trình tự gen chuyển nạp/Số lƣợng trình tự
gen đối chiếu) x 100
Bảng 4.17: Kết quả định lƣợng các mẫu thí nghiệm (2 lần lặp lại)
Kí hiệu mẫu
Kết quả định lƣợng (copy)
Tỷ lệ % chuyển
gen (%)
Giá trị trung bình (2 lần lặp lại)
p35S
Gen tham chiếu
82
82
B1
B2
B4
B6
B7
B10
B11
5940
945
7880
33,3
4900
7240
17700
96100
4460
12900
9270
43500
4270
331
6.18
21.19
61.09
0.36
11.26
169.56
5347.43
Một số nhận xét được rút ra như sau:
Các mẫu đều có sự hiện diện của p35S trong mẫu. Điều này phù hợp với kết quả
sàng lọc ỏ thí nghiệm trước.
Một số mẫu có sự hiện diện p35S trong mẫu khá thấp (B6: 33,3 copy), một số mẫu
hiện diện p35S rất lớn (B11: 17700 copy, B4: 7880 copy). Điều này có lẽ do sự khác
biệt về nguồn gốc các sản phẩm trong mẫu
Độ lệch chuẩn chu kỳ phát hiện của các mẫu không lớn. Điều này chứng tỏ, kết quả
định lượng không có sự biến động lớn ở các lần lặp lại.
Kết quả so sánh tỷ lệ phần trăm chuyển gen 3 mẫu B1, B2, B4 (đều là hạt giống bắp)
cho thấy sự hiện diện rất chênh lệch số lượng p35S trong các giống bắp khác nhau.
Tuy nhiên, dựa trên cơ sở lý thuyết, chúng tôi cho rằng chỉ có mẫu B1 là có kết quả
định lượng đáng tin cậy.
Mẫu B6 có kết quả định lượng khá tin cậy, dựa trên các kết quả thu nhận được ở
Bảng 4.17, 4.18 và 4.19. Các kết quả này chứng tỏ sự hiện diện của sản phẩm biến
đổi gen trong bắp trái ăn tươi tại Tp Hồ Chí Minh và phụ cận.
Tương tự, kết quả định lượng ở mẫu B7 cũng khá tin cậy, dựa trên các kết quả thu
nhận được. Mẫu này có nguồn gốc từ các loại bắp được trồng từ giống B1, B2, B4 và
các giống khác, sau đó qua quá trình xay xát tạo nên. Điều này cũng chứng minh có
sự hiện diện của hạt giống bắp chuyển gen tại Việt Nam.
Hai mẫu B10 và B11 có kết quả định lượng p35S vượt quá kết quả định lượng gen
tham chiếu. Điều này có thể do các mẫu này là sản phẩm chế biến, được tạo thành từ
rất nhiều thành phần nguyên liệu khác nhau, nên có thể con số này là tổng số lượng
promoter 35S trong mẫu chứ không chỉ riêng từ bắp chuyển gen. Mặc khác, do bắp
83
83
chỉ chiếm một tỷ lệ nhỏ trong thành phần nguyên liệu nên số lượng gen tham chiếu
phát hiện được rất thấp. Điều này đã ảnh hưởng đến kết quả thí nghiệm. Đây cũng
chính là nhược điểm của phương pháp định lượng dựa trên promoter 35S.
Như vậy, từ các kết quả thu nhận được từ thí nghiệm 5, chúng tôi rút ra một số kết luận như
sau:
- Phương pháp Real-Time PCR với mẫu dò Taqman có thể được ứng dụng cho việc định
lượng các mẫu sản phẩm biến đổi gen tại Việt Nam. Các kết quả thu nhận được từ các mẫu
có nguồn gốc khác nhau, từ các mẫu thô (hạt bắp) đến các mẫu đã trải qua quá trình chế
biến phức tạp (bột ngũ cốc dinh dưỡng) chứng tỏ khả năng áp dụng rộng rãi trên nhiều đối
tượng của phương pháp Real-Time PCR.
- Các kết quả trong thí nghiệm này cùng với kết quả sàng lọc bằng phương pháp Real-Time
PCR (SYBR Green I) cũng cố thêm chứng minh về độ nhạy cao của phương pháp này. Mặc
dù các kết quả là khác biệt, chúng cho thấy phương pháp Real-Time PCR có thể cho phép
phát hiện và định lượng trình tự DNA trong các mẫu có nguồn gốc và chất lượng rất khác
nhau.
- Các sai lệch về kết quả định lượng trong thí nghiệm này có thể giải thích do một số nguyên
nhân như sau:
Sự ngoại nhiễm do môi trường thực hiện thí nghiệm cũng như thao tác của kĩ thuật
viên gây ra. Các sai sót này có thể do việc bơm hút mẫu, môi trường làm việc không
vô trùng, dụng cụ thí nghiệm chưa khử trùng kỹ lưỡng…điều này chứng tỏ độ nhạy
rất cao của phương pháp đồng thời đặt ra các yêu cầu về việc chuẩn hóa các phòng
thí nghiệm định lượng.
Chất lượng DNA ly trích. Các kết quả khác biệt có thể xảy ra ngay trong cùng một
mẫu do chất lượng DNA ly trích có thể không ổn định hay do thao tác khi bơm hút
mẫu của kĩ thuật viên. Điều này đặt ra yêu cầu về việc hoàn thiện hóa các quy trình
ly trích DNA chuẩn nhằm đảm bảo tốt nhất chất lượng của DNA, đối tương chính
của thí nghiệm này.
Như vậy, tuy phương pháp này đặt ra một số yêu cầu đối với đầu tư trang thiết bị, chuẩn hóa
phòng thí nghiệm, huấn luyện kĩ thuật viên, khả năng ứng dụng thành công phương pháp này tại nước
ta là rất hứa hẹn.
84
84
KẾT LUẬN VÀ ĐỀ NGHỊ
1. Kết luận
Vấn đề định lượng sản phẩm biến đổi gen tại Việt Nam còn khá mới mẻ. Trong phạm vi khóa
luận này, chúng tôi chỉ mới bước đầu nghiên cứu về tính khả thi của phương pháp Real-Time PCR
trong việc định lượng DNA. Từ các kết quả thu nhận được, chúng tôi có một số kết luận về quy trình
định lượng các sản phẩm biến đổi gen như sau:
- Bước 1: Ly trích DNA mẫu sản phẩm.
Phương pháp đề nghị: Quy trình 3 (quy trình biến đổi). Tác nhân biến tính màng là
CTAB, biến tính protein là phenol/chloroform, tủa bằng Isopropanol trong điều kiện
lạnh (4
o
C).
Khó khăn: Lượng DNA thu được chưa cao, một số mẫu không ly trích được DNA.
Giải pháp: Thử nghiệm các phương pháp mới, sử dụng bộ kit ly trích.
- Bước 2: Sàng lọc sản phẩm biến đổi gen dựa trên promoter 35S.
Phương pháp đề nghị: Real-Time PCR với thuốc nhuộm SYBR Green I.
Khó khăn: Xuất hiện hiện tượng mồi bắt cặp không đặc hiệu trong một số mẫu.
Giải pháp: Cần tiến hành nghiên cứu tối ưu hóa phương pháp.
- Bước 3: Định lượng các sản phẩm biến đổi gen có kết quả sàng lọc dương tính, dựa
trên promoter 35S.
Phương pháp đề nghị: Real-Time PCR với mẫu dò đặc hiệu Taqman với độ nhạy 1
copy gen đích trong mẫu thí nghiệm .
Khó khăn: Chỉ có thể định lượng tổng số thành phần chuyển gen trong các mẫu hỗn
hợp.
Giải pháp: Cần định lượng các trình tự đặc hiệu hơn cho từng loại sản phẩm.
Về việc ứng dụng phương pháp Real-Time PCR khảo sát một số mẫu trên thị trường, chúng tôi
có một số kết luận sau:
- Các mẫu đã sàng lọc và định lượng có sản phẩm biến đổi gen: 3 mẫu hạt giống bắp, 1
mẫu bắp trái ăn tươi, 1 mẫu bột bắp làm nguyên liệu thức ăn gia súc, 1 mẫu thức ăn gia súc,
1 mẫu bột ngũ cốc dinh dưỡng.
85
85
- Có hiện diện các sản phẩm chuyển gen là thực phẩm cho người như: bắp trái ăn tươi,
bột ngũ cốc dinh dưỡng. Chúng có thể bắt nguồn hay có chứa thành phần nguyên liệu có
nguồn gốc từ cây trồng biến đổi gen.
2. Đề nghị
Tuy nhiên, do giới hạn thời gian và điều kiện thí nghiệm, cũng như đây là nghiên cứu ban đầu
về phương pháp Real-Time PCR trong định lượng sản phẩm biến đổi gen, chúng tôi có một số đề nghị
sau:
- Khóa luận chưa kiểm chứng độ đặc hiệu của mồi tự thiết kế và trong bộ kit định lượng
p35S vì vậy, cần tiến hành một số phương pháp khẳng định lại sản phẩm khuếch đại DNA
là p35S như: dùng enzyme cắt giới hạn, giải trình tự gen.
- Các mẫu âm tính có thể là mẫu chuyển gen bằng các thành phần khác (promoter 35S
chỉ hiện diện trong 70 % tổng số cây trồng biến đổi gen trên thế giới), vì vậy cần mở rộng
nghiên cứu sang các trình tự khác, làm phong phú thêm khả năng phát hiện và định lượng
- Tiến hành các nghiên cứu sâu hơn về định lượng sản phẩm biến đổi gen, tiến đến định
lượng các trình tự đặc hiệu của từng sản phẩm biến đổi gen, như: gen chuyển nạp, trình tự
nối promoter và gen chuyển nạp…nhằm khắc phục nhược điểm của phương pháp định
lượng dựa trên promoter 35S là chỉ có thể định lượng được tổng số thành phần biến đổi gen
trong các mẫu sản phẩm hỗn hợp.
- Tiến hành nghiên cứu và khảo sát trên số lượng mẫu lớn, các chủng loại mẫu khác
nhau để có tổng kết về tình hình sản phẩm biến đổi gen tại Việt Nam.
86
86
TÀI LIỆU THAM KHẢO
Tiếng Việt
1. Bùi Lan Anh, 2004. Khảo sát qui trình biến nạp gen trên cây thuốc lá (Nicotiana tabacum L.
cv Samsun). Khóa luận cử nhân khoa học, ĐH Khoa Học Tự Nhiên, Tp Hồ Chí Minh, Việt
Nam, trang 5-12, 16-19.
2. Lê Thanh Bình, 2004. Quản lý an toàn sinh học đối với sinh vật biến đổi gen và sản phẩm của
chúng. Hội nghị National Workshop on Risk Assessment of GM Crops-VN, tháng 10 năm 2004,
42 slide.
3. Clive James, 2004. Tình trạng cây trồng chuyển gen/cây trồng công nghệ sinh học được trồng
và mua bán trên toàn thế giới trong năm 2004. Báo cáo đánh giá. ISAAA, Mỹ, 14 trang.
4. Cơ quan dịch vụ quốc tế về tiếp thu các ứng dụng Công nghệ sinh học trong nông nghiệp
(ISAAA), 2005. Cây trồng công nghệ sinh học trên toàn cầu gần đạt mức tăng trưởng kỉ lục.
ISAAA, Mỹ. 2 trang.
5. Bùi Văn Lệ, 2004. Sinh vật biến đổi gen: Thực trạng và giải pháp. Báo cáo chuyên đề, ĐH
Khoa Học Tự Nhiên, Tp Hồ Chí Minh, Việt Nam, 55 slides.
6. La Tuấn Nghĩa, 2004. Bài phát biểu giới thiệu. Hội nghị National Workshop on Risk
Assessment of GM Crops-VN, tháng 10 năm 2004, 5 trang.
7. Nguyễn Hồng Minh Ngọc, 2004. Hoàn thiện phương pháp chuẩn đoán nông sản chuyển gen
dựa trên kỹ thuật PCR. Khóa luận Kỹ sư Nông nghiệp, Khoa Nông học, ĐH Nông Lâm Tp Hồ
Chí Minh, 56 trang.
8. Nguyễn Thái Thủy, 2004. PCR và Real-Time PCR. Bài giảng ĐH Nông Lâm, Tp Hồ Chí
Minh, Việt Nam, 110 slides.
9. Phạm Thị Anh Thư, 2004. “Giới thiệu về sinh vật chuyển đổi gen (GMOs)”. Hội thảo, Hội các
phòng thí nghiệm Việt Nam (VINATEST), Tp Hồ Chí Minh, Việt Nam.
Anh văn
10. Agrifood Awareness Australia Limited, 2004. Global GM Food Labelling Laws. Biotech
Bulletin 8. Agrifood Awareness Australia Limited, 7 pages.
11. Applied Biosystems. Real-Time PCR Vs. Traditional PCR. Tutorial, Applied Biosystems,
USA, 15 pages
12. Benbrook C., 2003. Impacts of Genetically Engineered Crops on Pesticide Use in the United
States: The First Eight Years. BioTech InfoNet, Technical Paper Number 6, USA, 46 pages.
87
87
13. Bio-Rad, 2005. GMO Testing in Food and Feed: Extraction, Detection and Quantification.
Bio-Rad, American, 8 pages.
14. Bio-Rad, 2005. MyiQ™ Single-Color Real-Time PCR Detection System. Instruction manual,
catalog number 170-9740. Bio-Rad, USA, 133 pages.
15. Bio-Rad. Biotechnology Explorer™ GMO Investigator™ Kit. Bio-Rad, USA, 79 pages.
16. Carolina Roa-Rodríguez, 2003. Promoters used to regulate gene expression. Cambia
Intellectual Property Resource, Australia, p. 1-24, 55-65.
17. Choi Yang Do, Cheong Jong-Joo, 2004. The Current Status of GMOs Development. Crop
Functional Genomics Center, School of Agricultural Biotechnology, Seoul National
University, p 3, 7-17.
18. Clive James, 2004. Biotech crop Countries and Mega-Countries, 2004. ISAAA, USA.
19. Clive James, 2004. Global Adoption rates (%) for pricipal Biotech Crops (Million Hectares).
ISAAA, USA.
20. Clive James, 2004. Global Area of Biotech Crops Million Hectares (1996 to 2004). ISAAA,
USA.
21. Clive James, 2005. Global Status of Commercialized Biotech/GM Crops: 2004. ISAAA, 12
pages.
22. Council For Biotechnology Information, 2002. Benefits of Plant Biotechnology. Council For
Biotechnology Information. 54 slides.
23. EEA (European Environment Agency), 1999. Genetically modified organisms. Environment in
the European Union at the turn of the century. Environmental assessment report No 2: 245-
261. EEA (European Environment Agency), EU.
24. Eyquem F., 2004. Quantitative Detection of Roundup Ready Soybean by ELISA. The Analysis
Of Food Samples For The Presence Of Genetically Modified Organisms (Querci M., Van den
Eede G., Jermini M.). European Commission Joint Research Center, EU. 21 pages.
25. Francisco Xavier Moreano Guerra, 2005. Development of techniques for the quantification of
DNA from genetically modified organisms in processed foods. Doctor thesis, Technology
University of Munich, Germany. 124 pages.
26. Friends of the Earth International, 2003. Biosafety Protocol ratification provides new measures
to guard against genetically modified organisms. Fact sheet, Friends of the Earth International,
3 pages.
27. Friends of the Earth, 2004. Genetically Modified Crops: a decade of failure [1994 - 2004].
Issue 105. Friends of the Earth International, 52 pages.
88
88
28. Gaskell George et at., 2000. Biotechnology and the European public. Nature Biotechnology 18:
935-938. Nature, American.
29. GeneScan. Calibration DNA Standards for 35S Corn DNA Quantification. Standard Lot
#0220502. GeneScan, Germany, 1 page.
30. GeneScan. GMOQuant 35S Screen Corn Kit. Protocol for use of Product cat. No. 5121200610.
GeneScan, Germany, 3 pages.
31. ISAAA. Does the 35S Promoter, which is viral in origin, have any potential to induce new
viruses?. ISAAA, USA, 21 slides.
32. Karudapuram Surekha, and Batey David. Detection of Genetically Modified Soybean in
Processed Foods Using Real-Time Quantitative PCR with SYBR Green I Dye on the DNA
Engine Opticon® 2 System. Application Note Vol.2, No.6, MJ Research, Inc., South San
Francisco, CA, USA, 4 pages.
33. Querci M., Maretti M., Mazzara M., 2004. Qualitative Detection of Mon810, Bt-176 Maize ang
Roundup Ready Soybean by PCR. The Analysis Of Food Samples For The Presence Of
Genetically Modified Organisms (Querci M., Van den Eede G., Jermini M.). European
Commission Joint Research Center, EU. p. 12.
34. Querci M., Mazzara M., 2004. Characteristics of the Qualitative PCR Systems Described in the
Manual. The Analysis Of Food Samples For The Presence Of Genetically Modified Organisms
(Querci M., Van den Eede G., Jermini M.). European Commission Joint Research Center, EU.
p. 4.
35. Querci M., Van den Eede G., Jermini M., 2004. Overview, General Introduction on Genetically
Modified Organisms (GMOs), EU legislation. The Analysis Of Food Samples For The
Presence Of Genetically Modified Organisms (Querci M., Van den Eede G., Jermini M.).
European Commission Joint Research Center, EU. p. 3-12, Annex 1.
36. Rogers S.O. and Bendich A.J, 1988. Extraction of DNA from plant tissues. Plant Molecular
Biology Manual A6, p.1-10.
37. Soil Association, 2004. GM Food: Scientific Evidence of Health Risks. Information sheet, Soil
Association, England, 3 pages.
38. Somma M., 2004. Extraction and Purification of DNA. The Analysis Of Food Samples For The
Presence Of Genetically Modified Organisms (Querci M., Van den Eede G., Jermini M.).
European Commission Joint Research Center, EU. p. 3-16.
39. The European Commission DG Joint Research Centre, 2000. Validation of analytical methods
for the identification and determination of genetically modified organisms (GMOs) in food and
food ingredients. Final Report, The European Commission DG Joint Research Centre, p. 9-12.
89
89
40. Viljoen, C.D., Botha, G.M. and Chetty, L. GMO testing: A prerequisite for Identity
Preservation of non-GM crops. GMO Testing Facility, Department of Plant Science,
University of the Free State, 6 pages.
41. Vu Duc Quang, 2004. Current Status of GM crops and GM-derived products in Vietnam.
National Workshop on Risk Assessment of GM Crops-VN, Oct 2004, 40 slides.
42. Weighardt F., 2004. Quantitative PCR for the Detection of GMOs. The Analysis Of Food
Samples For The Presence Of Genetically Modified Organisms (Querci M., Van den Eede G.,
Jermini M.). European Commission Joint Research Center, EU. p. 3-18.
Web
43. AgriQuality GMO Services – Quantification.
44. Arne Holst-Jensen, 2001. 2.2 DNA-based methods. CHAPTER 2 GMO detection methods and
validation. A review, with notes on future needs and directions. National Veterinary Institute,
NORWAY
/>ng%20PCR%20review.htm
45. Food and Feed Safety. Community register of GM Food & Feed.
46. Genetic Transcription and Regulation and Control.
47. Harrison Richard. Sequence Detection Systems: Principles of Real-Time PCR and Taqman
Systems, PE Biosustems, Australia and New Zealand.
48. Real-Time PCR. 2004.
90
90
PHỤ LỤC
Phụ lục 1: Đệm trích 1.
- Tris/HCl 0,05 mol/l
- EDTA 0,05 mol/l
- SDS 30 g/l
- Chỉnh pH = 8 bằng NaOH 1M hoặc HCl 1M.
- Hấp tiệt trùng.
- Giữ 4
o
C sử dụng trong 6 tháng.
Phụ lục 2: Đệm trích CTAB 2%
- CTAB 20 g/l
- NaCl 1,4 mol/l
- Tris/HCl 0,1 mol/l
- EDTA 0,02 mol/l
- Chỉnh pH = 8 bằng NaOH 1M hoặc HCl 1M.
- Hấp tiệt trùng.
- Giữ 4oC sử dụng trong 6 tháng.
Phụ lục 3: Dung dịch CTAB tủa.
- CTAB 5 g/l
- NaCl 0,04 mol/l
- Chỉnh pH = 8 bằng NaOH 1M hoặc HCl 1M.
- Hấp tiệt trùng.
- Giữ 4oC sử dụng trong 6 tháng.
Phụ lục 4: Dung dịch RNase A.
- RNase A 10 mg/ml
- Hòa tan trong nước cất siêu sạch.
- Giữ -20oC, chỉ nên sử dụng một lần sau khi rã đông.
Phụ lục 5: Dung dịch NaCl 1,2 M.
- NaCl 1,2 mol/l
- Hòa tan trong nước cất siêu sạch.
- Hấp tiệt trùng.
- Bảo quản ở nhiệt độ phòng.